[의학통계방법론] Ch17. Simple Linear Regression
Simple Linear Regression
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("dplyr")
library("kableExtra")
library("ggplot2")
library("chemCal")
library("broom")
17장
EXAMPLE 17.1

#데이터셋
age <- c(3.0,4.0,5.0,6.0,8.0,9.0,10.0,11.0,12.0,14.0,15.0,16.0,17.0)
wing_length<-c(1.4,1.5,2.2,2.4,3.1,3.2,3.2,3.9,4.1,4.7,4.5,5.2,5.0)
ex17_1 <- data.frame(age,wing_length)
ex17_1 %>%
kbl(caption = "Dataset of Example 17.1") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| age | wing_length |
|---|---|
| 3 | 1.4 |
| 4 | 1.5 |
| 5 | 2.2 |
| 6 | 2.4 |
| 8 | 3.1 |
| 9 | 3.2 |
| 10 | 3.2 |
| 11 | 3.9 |
| 12 | 4.1 |
| 14 | 4.7 |
| 15 | 4.5 |
| 16 | 5.2 |
| 17 | 5.0 |
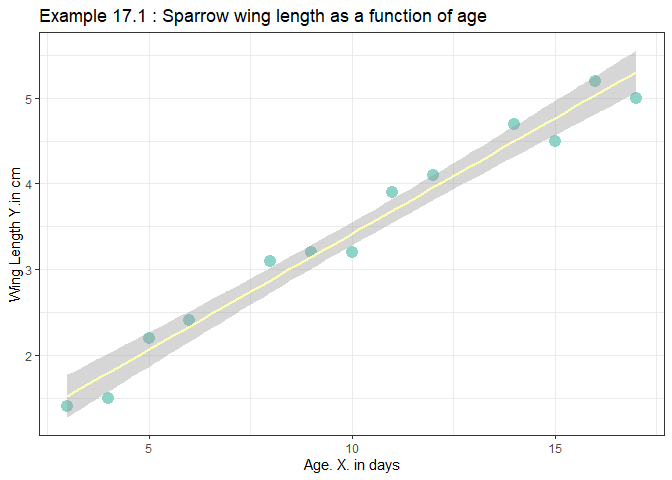
- 해당 데이터는 참새의 나이와 날개의 길이를 측정한 데이터이다.
- 이 데이터를 사용하여 연령과 날개 길이의 회귀직선을 그려보도록 한다.
library("ggplot2")
ggplot(ex17_1, aes(x=age, y=wing_length)) +
geom_point(color="#8dd3c7",size=4) +
stat_smooth(method = 'lm', color="#ffffb3")+
ggtitle("Example 17.1 : Sparrow wing length as a function of age")+
ylab("Wing Length Y.in cm")+
xlab("Age. X. in days")+
theme_bw()
EXAMPLE 17.2

- EXAMPLE 17.1 데이터로 Machine formula를 사용하여 회귀식을 구하도록 한다.
f_17_2 <- function(x){
n <- nrow(x)
sum_x <- sum(x[,1])
mean_x <- mean(x[,1])
sum_y <- sum(x[,2])
mean_y <- mean(x[,2])
sum_x2 <- sum(x[,1]^2)
sum_xy <- sum(x[,1]*x[,2])
b <- (sum_xy-((sum_x*sum_y)/n))/(sum_x2-(sum_x^2)/n)
a <- mean_y - b*mean_x
out <- data.frame(n,sum_x,mean_x,sum_y,mean_y,sum_x2,sum_xy,b,a)
return(out)
}
f_17_2(ex17_1)%>%
kbl(caption = "Result of simple linear regression equation",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| n | sum_x | mean_x | sum_y | mean_y | sum_x2 | sum_xy | b | a |
|---|---|---|---|---|---|---|---|---|
| 13 | 130 | 10 | 44.4 | 3.415385 | 1562 | 514.8 | 0.270229 | 0.7130945 |
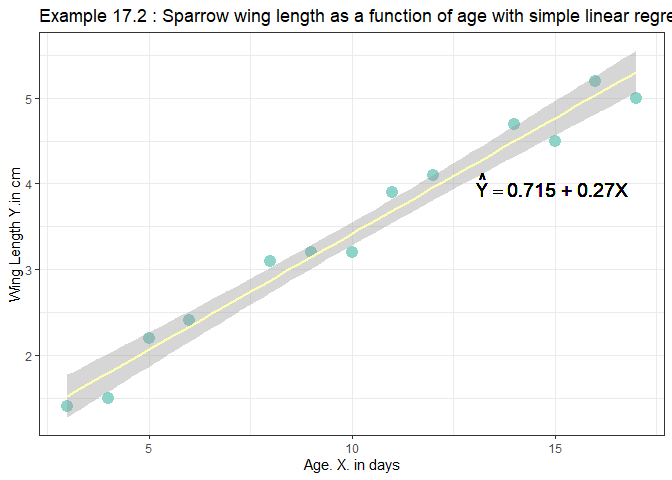
- 회귀식을 추정한 결과 \(\hat Y = 0.715+0.270X\) 로 도출되었다.
- Example 17.1에서 구한 회귀직선에 식을 넣으면 다음과 같다.
ggplot(ex17_1, aes(x=age, y=wing_length)) +
geom_point(color="#8dd3c7",size=4) +
stat_smooth(method = 'lm', color="#ffffb3")+
ggtitle("Example 17.2 : Sparrow wing length as a function of age with simple linear regression equation")+
ylab("Wing Length Y.in cm")+
xlab("Age. X. in days")+
theme_bw()+
geom_text(x=15, y=4, label=expression(hat(Y) == paste(0.715," + ",0.270,X, " ",)), cex = 5)
EXAMPLE 17.3
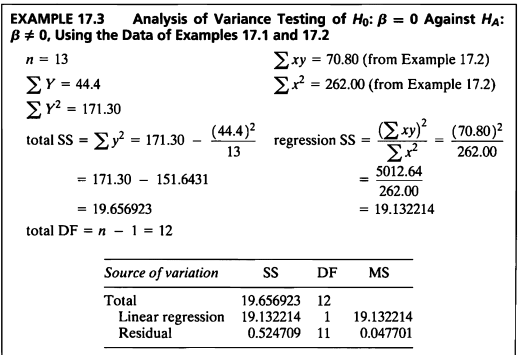

- 앞서 구한 회귀식의 ANOVA table을 작성하여 추정한 회귀식의 유의성을 판단하여 보겠다.
library("broom")
lm17_2 <- lm(wing_length~age,data=ex17_1)
aov17_2 <- anova(lm17_2)
aov17_2 %>% tidy() %>%
rename(" "="term","Sum Sq"="sumsq","Mean Sq"="meansq","F value"="statistic","Pr(>F)"="p.value") %>%
kable(caption = "ANOVA table",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| df | Sum Sq | Mean Sq | F value | Pr(\>F) | |
|---|---|---|---|---|---|
| age | 1 | 19.1322137 | 19.1322137 | 401.0875 | 0 |
| Residuals | 11 | 0.5247093 | 0.0477008 | NA | NA |
summ <- summary(lm17_2)
data.frame(summ$r.squared,summ$adj.r.squared) %>%
rename("R square"="summ.r.squared","Adjusted R square"="summ.adj.r.squared") %>%
kable(caption = "R square",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| R square | Adjusted R square |
|---|---|
| 0.9733066 | 0.97088 |
- 회귀직선의 유의성을 판단한 결과, F값은 401.09, p-value<0.001 이므로 \(H_0: \beta_1=0\)은 기각되었다.
따라서 유의수준 5%하에 회귀직선은 유의하다고 할 수 있다. - 결정계수 값은 0.97로, 총변동중에서 회귀직선에 의해 설명되는 부분이 97%임을 확인하였다.
EXAMPLE 17.4

- EXAMPLE 1,2에 사용된 데이터를 가지고 회귀계수의 유의성 검정을 하기 위해 t-test를 진행해 보도록 하겠다.
직접 함수를를 작성하여 구하여 보겠다.
f_17_4 <- function(x){
alpha <- 0.05
n <- nrow(x)
estimate <- f_17_2(x)$b
r2 <- summ$r.squared
syx2 <- (sd(x[,2])*sqrt((1-r2)*(13-1)/(13-2)))^2
sum_x2 <- sum(x[,1]^2)-(sum(x[,1])^2)/n
se <- sqrt(syx2/sum_x2)
t <- estimate/se
crit <- qt(1-alpha/2,n-2)
pvalue <- pt(t,n-2,lower.tail = F)
if(pvalue < 0.05){
Reject_H0 <- "yes"
}else{
Reject_H0 <- "No"
}
out <- data.frame(n,estimate,se,t,pvalue,Reject_H0)
return(out)
}
f_17_4(ex17_1)%>%
kbl(caption = "Result of t test",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| n | estimate | se | t | pvalue | Reject_H0 |
|---|---|---|---|---|---|
| 13 | 0.270229 | 0.0134931 | 20.02717 | 0 | yes |
R에 내장된 함수로 구하여 보겠다.
summ$coefficients %>%
as.data.frame() %>%
kable(caption = "Coefficient",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Estimate | Std. Error | t value | Pr(\>\|t\|) | |
|---|---|---|---|---|
| (Intercept) | 0.7130945 | 0.1479045 | 4.821319 | 0.0005347 |
| age | 0.2702290 | 0.0134931 | 20.027169 | 0.0000000 |
- 본 검정의 가설은 다음과 같다.
- 회귀계수의 유의성 검정 결과 age에 대한 coefficient 값은 0.2702290, t 값은 20.02 이며 p-value<0.001 로써 유의수준 0.05보다 작아 귀무가설을 기각할 충분한 근거를 가진다.
-그러므로 유의수준 5%하에 회귀계수는 유의하다고 할 수 있다.
EXAMPLE 17.5


- 다음의 식을 통하여 표준오차를 구해해 신뢰구간을 예측해보도록 하겠겠다.
- 13 day old의 새들의 날개의 길이의 모평균은 얼마인지 표준오차를 구해 신뢰구간을 계산해보도록 하겠다.
f_17_5 <- function(x,param){
alpha <- 0.05
n <- nrow(x)
b <- f_17_2(x)$b
a <- f_17_2(x)$a
yi <- a+b*param
r2 <- summ$r.squared
mean_x <- mean(x[,1])
syx2 <- (sd(x[,2])*sqrt((1-r2)*(13-1)/(13-2)))^2
sum_x <- sum(x[,1])
sum_x2 <- sum(x[,1]^2)
sumx2 <- sum_x2-(sum_x^2)/n
syi <- sqrt(syx2*((1/param)+((param-mean_x)^2/(sum_x2-(sum_x^2)/n))))
syi_10 <- sqrt(syx2*((1/10)+(1/param)+((param-mean_x)^2/(sum_x2-(sum_x^2)/n))))
syi_1 <- sqrt(syx2*(1+(1/param)+((param-mean_x)^2/(sum_x2-(sum_x^2)/n))))
crit <- qt(1-alpha/2,n-2)
L1_1 <- yi-crit*syi
L2_1 <- yi+crit*syi
L1_2 <- yi-crit*syi_10
L2_2 <- yi+crit*syi_10
L1_3 <- yi-crit*syi_1
L2_3 <- yi+crit*syi_1
out <- data.frame(yi,syi,L1_1,L2_1,syi_10,L1_2,L2_2,syi_1,L1_3,L2_3)
return(out)
}
f_17_5(ex17_1,13)%>%
kbl(caption = "Result",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| yi | syi | L1_1 | L2_1 | syi_10 | L1_2 | L2_2 | syi_1 | L1_3 | L2_3 |
|---|---|---|---|---|---|---|---|---|---|
| 4.226072 | 0.0728552 | 4.065719 | 4.386425 | 0.100389 | 4.005117 | 4.447026 | 0.2302362 | 3.719325 | 4.732818 |
- Equation a)를 사용하였을 때 13일 살의 새의 날개 길이의 모평균의 95% 신뢰구간은 [4.065, 4.386] 이며,
b)를 사용했을 떄는 [4.005, 4.445] 그리고,
c)를 사용했을 때는 [3.719, 4.731]이다.
EXAMPLE 17.6

- Estimated Y Value에 대하여 검정을 진행하여 보겠겠다.
f_17_6 <- function(x,param,compare){
alpha <- 0.05
n <- nrow(x)
b <- f_17_2(x)$b
a <- f_17_2(x)$a
yi <- a+b*param
r2 <- summ$r.squared
mean_x <- mean(x[,1])
syx2 <- (sd(x[,2])*sqrt((1-r2)*(13-1)/(13-2)))^2
sum_x <- sum(x[,1])
sum_x2 <- sum(x[,1]^2)
sumx2 <- sum_x2-(sum_x^2)/n
syi <- sqrt(syx2*((1/param)+((param-mean_x)^2/(sum_x2-(sum_x^2)/n))))
t <- (yi-compare)/syi
crit <- qt(1-alpha/2,n-2)
pvalue <- pt(t,n-2,lower.tail = F)
if(pvalue < 0.05){
Reject_H0 <- "yes"
}else{
Reject_H0 <- "No"
}
out <- data.frame(yi,t,crit,pvalue,Reject_H0)
return(out)
}
f_17_6(ex17_1,param=13,compare=4)%>%
kbl(caption = "Result",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| yi | t | crit | pvalue | Reject_H0 |
|---|---|---|---|---|
| 4.226072 | 3.103028 | 2.200985 | 0.0050248 | yes |
- 예측된 Y값 검정 결과 검정통계량 t값은 3.10이고 p-value=0.005 이므므로 유의수준 0.05보다 작아 귀무가설을 기각할 충분한 근거가 있다.
- 그러므로 유의수준5%하에 새들의 날개의 길이에 대한 모평균이 4cm 보다 크다는 충분한 근거가 있다고 할 수 있다.
EXAMPLE 17.7

- 이번에는 y값이 주어졌을 때 x값을 구해보도록 하겠다.
- 즉, 날개의 길이가 주어졌을 때, 새들의 나이를 예측해보는 inverse에 관한 문제이다.
- 날개의 길이가 4.5cm 라고 했을 때 신뢰구간을 구하는 방식은 다음과 같다.
R의 chemCal 패키지의 inverse.predict() 함수를 사용하여 구해보겠다.
library("chemCal")
inverse_result <- inverse.predict(lm17_2,4.5)
inverse_result <- as.data.frame(inverse_result)
inverse_result[2,1:3] <- "."
inverse_result %>%
kbl(caption = "Result of Inverse Prediction",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Prediction | Standard.Error | Confidence | Confidence.Limits |
|---|---|---|---|
| 14.0136897001304 | 0.862343975232319 | 1.89800629238076 | 12.11568 |
| . | . | . | 15.91170 |
- 위의 결과를 보아 날개의 길이가 4.5cm라고 주어졌을 때,
예상되는 새의 나이의 값은 14.013이고,
이에 대한 95% 신뢰구간은 [12.115, 15.911] 이다.
EXAMPLE 17.8a
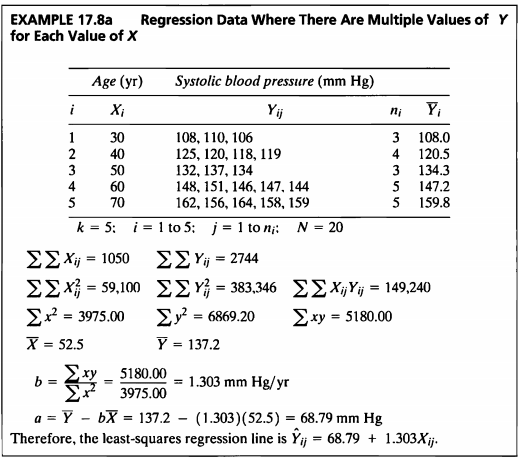
#데이터셋
age<-c(rep(30,3),rep(40,4),rep(50,3),rep(60,5),rep(70,5))
sbp<-c(108,110,106,125,120,118,119,132,137,134,148,151,146,147,144,162,156,164,158,159)
ex17_8 <- data.frame(age,sbp)
ex17_8 %>%
kbl(caption = "Dataset of Example 17_8a",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| age | sbp |
|---|---|
| 30 | 108 |
| 30 | 110 |
| 30 | 106 |
| 40 | 125 |
| 40 | 120 |
| 40 | 118 |
| 40 | 119 |
| 50 | 132 |
| 50 | 137 |
| 50 | 134 |
| 60 | 148 |
| 60 | 151 |
| 60 | 146 |
| 60 | 147 |
| 60 | 144 |
| 70 | 162 |
| 70 | 156 |
| 70 | 164 |
| 70 | 158 |
| 70 | 159 |
- 해당 데이터는 나이와 이에 따른 수축기혈압의 데이터이다.
- 각각의 x에 대해 multiple value Y를 가지는 회귀분석에 대한 문제이다.
\[\begin{aligned} H_0 &: Regression\ model\ is\ not\ significant \\ H_A &: Regression\ model\ is\ significant \end{aligned}\]모형의 유의성 검정
lm17_8 <- lm(sbp~age,data=ex17_8)
aov17_8 <- anova(lm17_8)
aov17_8%>% tidy() %>%
rename(" "="term","Sum Sq"="sumsq","Mean Sq"="meansq","F value"="statistic","Pr(>F)"="p.value") %>%
kable(caption = "ANOVA table",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| df | Sum Sq | Mean Sq | F value | Pr(\>F) | |
|---|---|---|---|---|---|
| age | 1 | 6750.2893 | 6750.289308 | 1021.819 | 0 |
| Residuals | 18 | 118.9107 | 6.606149 | NA | NA |
- 모형의 유의성 검정 결과 검정통계량 F값은 1021.819, p-value<0.001이므로 유의수준 0.05보다 작아 귀무가설을 기각할 충분한 근거를 가진다.
- 그러므로 유의수준 5%하에 회귀모형은 유의하다고 할 수 있다.
\[\begin{aligned} H_0 &: \beta=0 \\ H_A &: \beta \ne 0 \end{aligned}\]회귀계수의 유의성 검정
summary(lm17_8)[4] %>%
as.data.frame %>%
rename("Estimate"="coefficients.Estimate","Standard Error"="coefficients.Std..Error","t value"="coefficients.t.value","Pr(>F)"="coefficients.Pr...t..") %>%
kable(caption = "Test of Coefficients",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Estimate | Standard Error | t value | Pr(\>F) | |
|---|---|---|---|---|
| (Intercept) | 68.784906 | 2.2160746 | 31.03907 | 0 |
| age | 1.303145 | 0.0407667 | 31.96590 | 0 |
- 회귀계수의 유의성 검정 결과 검정통계량 t값은 31.039, p-value<0.001이므로 유의수준 0.05보다 작아 귀무가설을 기각할 충분한 근거를 갖는다.
- 그러므로 유의수준 5%하에 회귀계수는 유의하다고 할 수 있다.
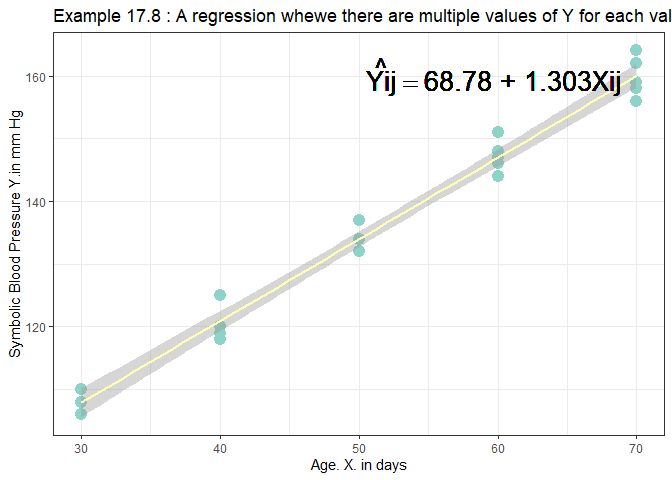
- 추정된 회귀식은 \(\hat Y_{ij}=68.78+1.303X_{ij}\) 이다.
- 회귀선을 그려보면 다음과 같다.
ggplot(ex17_8, aes(x=age, y=sbp)) +
geom_point(color="#8dd3c7",size=4) +
stat_smooth(method = 'lm', color="#ffffb3")+
ggtitle("Example 17.8 : A regression whewe there are multiple values of Y for each value of X")+
ylab("Symbolic Blood Pressure Y.in mm Hg")+
xlab("Age. X. in days")+
theme_bw()+
geom_text(x=60, y=160, label=expression(hat(Yij) == paste(68.78," + ",1.303,Xij, " ",)), cex = 7)
EXAMPLE 17.8b


- EXAMPLE 17.8a에서 구한 회귀모형의 선형성 검정을 위해 적합결여 F-test 를 수행하도록 하겠다.
- 선형성 검정을 하기 위해서는 단순 선형모형을 고려한 축소모형과 설명변수 각각의 값들을 factor화 하여 적합한 모형인 전체모형을 구한 뒤 Reduced model과 Full model 사이의 분산분석을 통하여 선형성을 검정한다.
Reduced model
rm <- lm(sbp~age,data=ex17_8)
summary(rm) %>% tidy %>%
rename("t value"="statistic","P value"="p.value") %>%
kable(caption = "Reduced model",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | estimate | std.error | t value | P value |
|---|---|---|---|---|
| (Intercept) | 68.784906 | 2.2160746 | 31.03907 | 0 |
| age | 1.303145 | 0.0407667 | 31.96590 | 0 |
Full model
fm <- lm(sbp~factor(age),data=ex17_8)
summary(fm) %>% tidy %>%
rename("t value"="statistic","P value"="p.value") %>%
kable(caption = "Reduced model",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | estimate | std.error | t value | P value |
|---|---|---|---|---|
| (Intercept) | 108.00000 | 1.614288 | 66.902558 | 0.00e+00 |
| factor(age)40 | 12.50000 | 2.135502 | 5.853424 | 3.17e-05 |
| factor(age)50 | 26.33333 | 2.282948 | 11.534793 | 0.00e+00 |
| factor(age)60 | 39.20000 | 2.041931 | 19.197516 | 0.00e+00 |
| factor(age)70 | 51.80000 | 2.041931 | 25.368146 | 0.00e+00 |
\[\begin{aligned} H_0 &: The \ population \ regression \ is \ linear. \\ H_A &: The \ population \ regression \ is \ not \ linear. \end{aligned}\]선형성 검정 (Lack-of-fit test)
lof <- anova(rm,fm)
lof %>% tidy %>%
rename("Res df"="df.residual","RSS"="rss","Sum sq"="sumsq","F value"="statistic","P value"="p.value") %>%
kable(caption = "Reduced model",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | Res df | RSS | df | Sum sq | F value | P value |
|---|---|---|---|---|---|---|
| sbp \~ age | 18 | 118.9107 | NA | NA | NA | NA |
| sbp \~ factor(age) | 15 | 117.2667 | 3 | 1.644025 | 0.0700977 | 0.9750327 |
- 적합결여 검정 결과 검정통계량 F값은 0.07, p-value=0.975 이므로 유의수준 0.05보다 커 귀무가설을 기각할 충분한 근거를 가지고있지 않다.
- 그러므로 유의수준 5%하에 모 회귀모델은 선형성을 따른다고 할 충분한 근거가 있다고 할 수 있다.
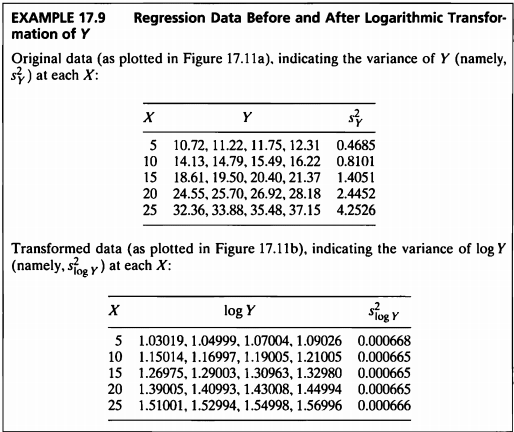
EXAMPLE 17.9
#데이터셋
X<-c(rep(5,4),rep(10,4),rep(15,4),rep(20,4),rep(25,4))
Y<-c(10.72,11.22,11.75,12.31,14.13,14.79,15.49,16.22,18.61,19.50,20.40,21.37, 24.55,25.70,26.92,28.18,32.36,33.88,35.48,37.15)
ex17_9 <- data.frame(X,Y)
ex17_9%>%
kbl(caption = "Dataset of Example 17_9",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| X | Y |
|---|---|
| 5 | 10.72 |
| 5 | 11.22 |
| 5 | 11.75 |
| 5 | 12.31 |
| 10 | 14.13 |
| 10 | 14.79 |
| 10 | 15.49 |
| 10 | 16.22 |
| 15 | 18.61 |
| 15 | 19.50 |
| 15 | 20.40 |
| 15 | 21.37 |
| 20 | 24.55 |
| 20 | 25.70 |
| 20 | 26.92 |
| 20 | 28.18 |
| 25 | 32.36 |
| 25 | 33.88 |
| 25 | 35.48 |
| 25 | 37.15 |
- 원 데이터와 로그변환한 데이터 간의 차이를 확인해보도록 하겠다.
로그변환
log_y <- log(Y,base=10)
ex17_9_trans <- data.frame(X,log_y)
ex17_9_trans%>%
kbl(caption = "Logarithmic transformation Dataset of Example 17_9",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| X | log_y |
|---|---|
| 5 | 1.030195 |
| 5 | 1.049993 |
| 5 | 1.070038 |
| 5 | 1.090258 |
| 10 | 1.150142 |
| 10 | 1.169968 |
| 10 | 1.190051 |
| 10 | 1.210051 |
| 15 | 1.269746 |
| 15 | 1.290035 |
| 15 | 1.309630 |
| 15 | 1.329805 |
| 20 | 1.390051 |
| 20 | 1.409933 |
| 20 | 1.430075 |
| 20 | 1.449941 |
| 25 | 1.510009 |
| 25 | 1.529943 |
| 25 | 1.549984 |
| 25 | 1.569959 |
ex17_9_result1 <- lm(Y~X, data=ex17_9)
#ex17.9_result1$residuals
ex17_9_result2 <- lm(log_y~X, data=ex17_9_trans)
#ex17.9_result2$residuals
par(mfrow=c(1,2))
plot(X, Y, pch=16,las=1,xlab="X", ylab="Y",ylim=c(0,40),xlim=c(0,25), main="The original data", cex.main=1, col="#8f7450")
abline(coef(ex17_9_result1), lwd=2, col="orange")
plot(X,log_y,pch=16,las=1,xlab="X",ylab="Y",ylim=c(1.0,1.6),xlim=c(0,25), main="The data after transformation", cex.main=1, col="#8f7450")
abline(coef(ex17_9_result2), lwd=2, col="orange")
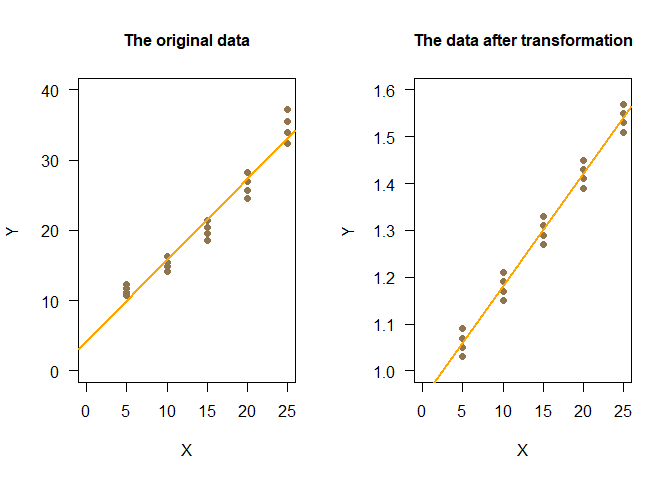
par(mfrow=c(1,2))
plot(X, ex17_9_result1$residuals, xlab="X", ylab="residuals", main = "The original data", cex.main=1, pch=16, col="#8dd3c7")
plot(X, ex17_9_result2$residuals, xlab="X", ylab="residuals", main = "The data after transformation", cex.main=1, pch=16, col="#8dd3c7")
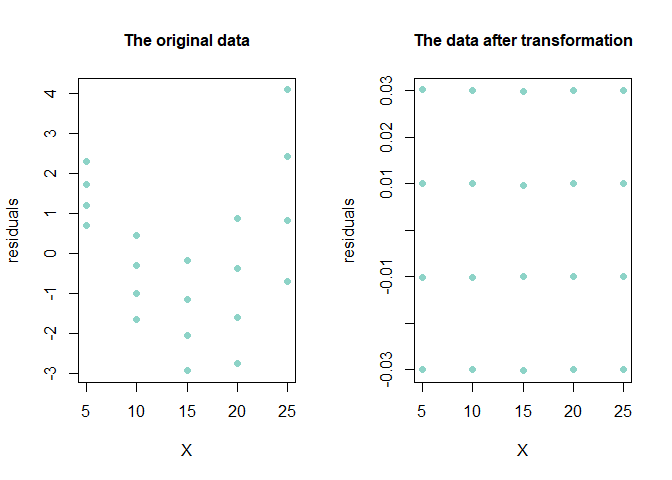
par(mfrow=c(1,2))
qqnorm(ex17_9_result1$residuals, main = "The original data", cex.main=1, pch=16, col="#8dd3c7")
qqnorm(ex17_9_result2$residuals, main = "The data after transformation", cex.main=1, pch=16, col="#8dd3c7")
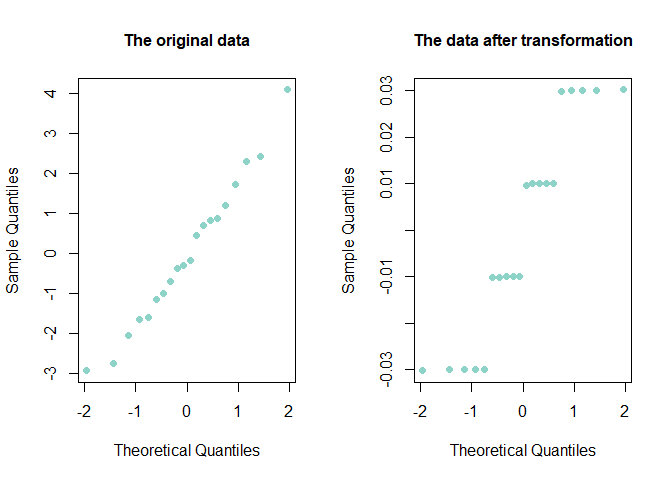
- 데이터를 로그변환하였을 때 더 강한 선형성을 가짐을 알 수 있다.
- 그리고 로그변환하였을 때의 잔차의 절대값이 변환 전보다 크게 줄었음을 알 수 있다.
origin <- c(var(Y[ex17_9$X==5]),var(Y[ex17_9$X==10]),var(Y[ex17_9$X==15]),var(Y[ex17_9$X==20]),var(Y[ex17_9$X==25]))
logtrans <- c(var(log_y[ex17_9_trans$X==5]),var(log_y[ex17_9_trans$X==10]),var(log_y[ex17_9_trans$X==15]),var(log_y[ex17_9_trans$X==20]),var(log_y[ex17_9_trans$X==25]))
variance <- data.frame(origin,logtrans)
variance%>%
kbl(caption = "Variance of each of original data set Logarithmic transformation",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| origin | logtrans |
|---|---|
| 0.4684667 | 0.0006682 |
| 0.8100917 | 0.0006654 |
| 1.4051333 | 0.0006652 |
| 2.4452250 | 0.0006654 |
| 4.2525583 | 0.0006659 |
SAS 프로그램 결과
SAS 접기/펼치기 버튼
17장
EXAMPLE 17.1
DATA ex17_1;
INPUT age wing @@;
CARDS;
3 1.4 4 1.5 5 2.2 6 2.4 8 3.1 9 3.2
10 3.2 11 3.9 12 4.1 14 4.7 15 4.5 16 5.2 17 5
;
RUN;
PROC PRINT DATA=ex17_1;
RUN;
| OBS | age | wing |
|---|---|---|
| 1 | 3 | 1.4 |
| 2 | 4 | 1.5 |
| 3 | 5 | 2.2 |
| 4 | 6 | 2.4 |
| 5 | 8 | 3.1 |
| 6 | 9 | 3.2 |
| 7 | 10 | 3.2 |
| 8 | 11 | 3.9 |
| 9 | 12 | 4.1 |
| 10 | 14 | 4.7 |
| 11 | 15 | 4.5 |
| 12 | 16 | 5.2 |
| 13 | 17 | 5.0 |
PROC SGPLOT DATA=ex17_1;
REG y=wing x=age;
INSET / border title="Wing Lengths of 13 Sparrows of Various Ages." position=topleft;
RUN;
EXAMPLE 17.2
ods graphics off;ods exclude all;ods noresults;
PROC REG DATA=ex17_1;
MODEL wing=age;
PLOT wing*age;
ODS OUTPUT ParameterEstimates=ParameterEstimates;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=ParameterEstimates;
RUN;
| OBS | Model | Dependent | Variable | DF | Estimate | StdErr | tValue | Probt |
|---|---|---|---|---|---|---|---|---|
| 1 | MODEL1 | wing | Intercept | 1 | 0.71309 | 0.14790 | 4.82 | 0.0005 |
| 2 | MODEL1 | wing | age | 1 | 0.27023 | 0.01349 | 20.03 | <.0001 |
EXAMPLE 17.3
ods graphics off;ods exclude all;ods noresults;
PROC REG DATA=ex17_1;
MODEL wing=age;
PLOT wing*age;
ODS OUTPUT ANOVA=ANOVA FitStatistics=FitStatistics;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=ANOVA;
RUN;
PROC PRINT DATA= FitStatistics label;
RUN;
| OBS | Model | Dependent | Source | DF | SS | MS | FValue | ProbF |
|---|---|---|---|---|---|---|---|---|
| 1 | MODEL1 | wing | Model | 1 | 19.13221 | 19.13221 | 401.09 | <.0001 |
| 2 | MODEL1 | wing | Error | 11 | 0.52471 | 0.04770 | _ | _ |
| 3 | MODEL1 | wing | Corrected Total | 12 | 19.65692 | _ | _ | _ |
| OBS | Model | Dependent | Label1 | cValue1 | nValue1 | Label2 | cValue2 | nValue2 |
|---|---|---|---|---|---|---|---|---|
| 1 | MODEL1 | wing | Root MSE | 0.21841 | 0.218405 | R-Square | 0.9733 | 0.973307 |
| 2 | MODEL1 | wing | Dependent Mean | 3.41538 | 3.415385 | Adj R-Sq | 0.9709 | 0.970880 |
| 3 | MODEL1 | wing | Coeff Var | 6.39475 | 6.394748 | 0 |
EXAMPLE 17.4
ods graphics off;ods exclude all;ods noresults;
PROC REG DATA=ex17_1;
MODEL wing=age;
ODS OUTPUT ParameterEstimates=PE;
RUN;
DATA pe1;
SET pe;
t=Estimate/StdErr;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=pe1;
VAR Estimate StdErr t;
WHERE Variable='age';
RUN;
| OBS | Estimate | StdErr | t |
|---|---|---|---|
| 2 | 0.27023 | 0.01349 | 20.0272 |
EXAMPLE 17.5
DATA ex17_5;
INPUT age @@;
CARDS;
13
;
RUN;
DATA ex17_5f;
SET ex17_5 ex17_1 ;
RUN;
ods graphics off; ods exclude all; ods noresults;
PROC REG DATA=ex17_5f;
MODEL wing=age / P CLM CLI ;
ODS OUTPUT OutputStatistics=OutputStatistics;
RUN;
ods graphics on; ods exclude none; ods results;
PROC PRINT DATA=OutputStatistics LABEL;
OPTIONS firstobs=1 obs=1;
VAR StdErrMeanPredict LowerCLMean UpperCLMean;
RUN;
| OBS | Std Err Mean Predict | Lower 95% CL Mean | Upper 95% CL Mean |
|---|---|---|---|
| 1 | 0.0729 | 4.0657 | 4.3864 |
EXAMPLE 17.6
PROC IML;
RESET nolog;
y=4.225;
s=0.073;
t=(y-4)/s;
p=1-probt(t,11);
PRINT t p;
RUN;
QUIT;
| t | p |
|---|---|
| 3.0821918 | 0.0052152 |
EXAMPLE 17.7
PROC IML;
USE ex17_1;
READ all;
CLOSE ex17_1;
Yi = 4.5;
df = 11;
NObsUsed = nrow(wing);
x = repeat(1, nrow(age)) || age;
y = wing;
n=nrow(x);
k=ncol(x);
xpx=x`*x;
xpy=x`*y;
xpxi=inv(xpx);
beta=xpxi*xpy;
sse = y`*y-xpy`*beta;
dfe = n-k;
mse = sse/dfe;
rmse = sqrt(mse);
rsquare = 1-sse/((y-y[:])[##]);
covb = mse#xpxi ;
std_beta = SQRT(VECDIAG(covb)) ;
sy = std(y);
Syx = sy * sqrt((1-rsquare)*(n-1)/(n-2));
S2yx = Syx **2;
xbar = mean(age);
Sxx = (age- xbar)`*(age- xbar);
Ybar=mean(y);
a = beta[1]; Sa=std_beta[1];
Byx = beta[2]; Sb=std_beta[2];
tvalue = tinv(0.975, df);
prediced_X = (Yi-a) / Byx;
D = Byx**2 - (tvalue**2 * Sb**2);
H = (tvalue / D) * sqrt ( S2yx * (D * (1+(1/NObsUsed)) + ((Yi - Ybar)**2) / Sxx));
LL = Xbar + ((Byx * (Yi - Ybar)) / D) - H;
UL = Xbar + ((Byx * (Yi - Ybar)) / D) + H;
PRINT prediced_X LL UL;
RUN;
QUIT;
| prediced_X | LL | UL |
|---|---|---|
| 14.01369 | 12.152556 | 15.972963 |
EXAMPLE 17.8
DATA ex17_8;
INPUT x y @@;
CARDS;
30 108 30 110 30 106
40 125 40 120 40 118 40 119
50 132 50 137 50 134
60 148 60 151 60 146 60 147 60 144
70 162 70 156 70 164 70 158 70 159
;
RUN;
PROC REG DATA=ex17_8;;
MODEL y=x / lackfit;
RUN;
The REG Procedure
Model: MODEL1
Dependent Variable: y
| Number of Observations Read | 20 |
|---|---|
| Number of Observations Used | 20 |
| Analysis of Variance | |||||
|---|---|---|---|---|---|
| Source | DF | Sum of Squares | Mean Square | F Value | Pr > F |
| Model | 1 | 6750.28931 | 6750.28931 | 1021.82 | <.0001 |
| Error | 18 | 118.91069 | 6.60615 | ||
| Lack of Fit | 3 | 1.64403 | 0.54801 | 0.07 | 0.9750 |
| Pure Error | 15 | 117.26667 | 7.81778 | ||
| Corrected Total | 19 | 6869.20000 | |||
| Root MSE | 2.57024 | R-Square | 0.9827 |
|---|---|---|---|
| Dependent Mean | 137.20000 | Adj R-Sq | 0.9817 |
| Coeff Var | 1.87336 |
| Parameter Estimates | |||||
|---|---|---|---|---|---|
| Variable | DF | Parameter Estimate | Standard Error | t Value | Pr > |t| |
| Intercept | 1 | 68.78491 | 2.21607 | 31.04 | <.0001 |
| x | 1 | 1.30314 | 0.04077 | 31.97 | <.0001 |
The REG Procedure
Model: MODEL1
Dependent Variable: y
EXAMPLE 17.9
DATA ex17_9 ;
INPUT x y @@ ;
CARDS;
5 10.72 5 11.22 5 11.75 5 12.31
10 14.13 10 14.79 10 15.49 10 16.22
15 18.61 15 19.50 15 20.40 15 21.37
20 24.55 20 25.70 20 26.92 20 28.18
25 32.36 25 33.88 25 35.48 25 37.15
;
RUN;
DATA ex17_9_log;
SET ex17_9;
log_y = log10(y);
RUN;
PROC REG DATA=ex17_9_log;
MODEL y=x;
ODS SELECT ResidualPlot;
RUN;
PROC REG DATA=ex17_9_log;
MODEL log_y=x;
ODS SELECT ResidualPlot;
RUN;
The REG Procedure
Model: MODEL1
Dependent Variable: y
The REG Procedure
Model: MODEL1
Dependent Variable: log_y
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
