[의학통계방법론] Ch16. Multivariate Analysis of Variance
Multivariate Analysis of Variance
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("foreign")
library("dplyr")
library("kableExtra")
library("mvoutlier")
library("mvnormtest")
library("broom")
library("biotools")
16장
16장 연습문제 불러오기
ex16_1 <- read.spss('Exam 16.1.sav', to.data.frame=T)
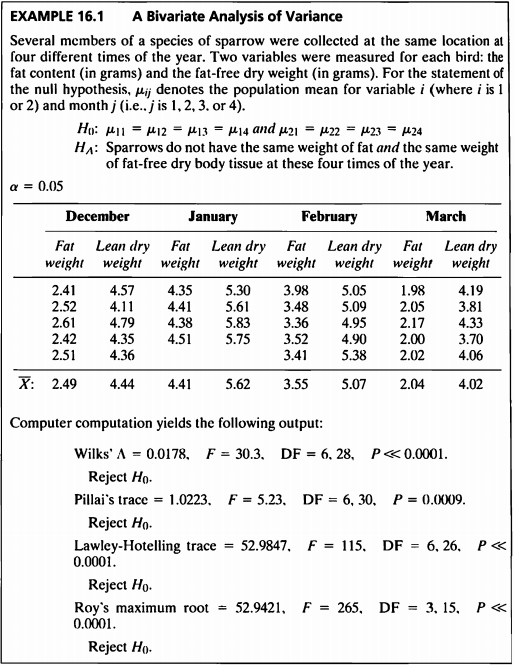
EXAMPLE 16.1
#데이터셋
ex16_1%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Y11 | Y12 | Y21 | Y22 | Y31 | Y32 | Y41 | Y42 |
|---|---|---|---|---|---|---|---|
| 2.41 | 4.57 | 4.35 | 5.30 | 3.98 | 5.05 | 1.98 | 4.19 |
| 2.52 | 4.11 | 4.41 | 5.61 | 3.48 | 5.09 | 2.05 | 3.81 |
| 2.61 | 4.79 | 4.38 | 5.83 | 3.36 | 4.95 | 2.17 | 4.33 |
| 2.42 | 4.35 | 4.51 | 5.75 | 3.52 | 4.90 | 2.00 | 3.70 |
| 2.51 | 4.36 | NA | NA | 3.41 | 5.38 | 2.02 | 4.06 |
- 분석을 위하여 데이터를 정제하도록 하겠다.
month <- c(rep("December",5),rep("January",5),rep("February",5),rep("March",5))
fat <- c(ex16_1$Y11,ex16_1$Y21,ex16_1$Y31,ex16_1$Y41)
lean <- c(ex16_1$Y12,ex16_1$Y22,ex16_1$Y32,ex16_1$Y42)
ex16 <- data.frame(month,fat,lean)
ex16%>%
kbl(caption = "Dataset of Example 16.1") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| month | fat | lean |
|---|---|---|
| December | 2.41 | 4.57 |
| December | 2.52 | 4.11 |
| December | 2.61 | 4.79 |
| December | 2.42 | 4.35 |
| December | 2.51 | 4.36 |
| January | 4.35 | 5.30 |
| January | 4.41 | 5.61 |
| January | 4.38 | 5.83 |
| January | 4.51 | 5.75 |
| January | NA | NA |
| February | 3.98 | 5.05 |
| February | 3.48 | 5.09 |
| February | 3.36 | 4.95 |
| February | 3.52 | 4.90 |
| February | 3.41 | 5.38 |
| March | 1.98 | 4.19 |
| March | 2.05 | 3.81 |
| March | 2.17 | 4.33 |
| March | 2.00 | 3.70 |
| March | 2.02 | 4.06 |
- 해당 데이터는 참새들의 지방 함량를 측정한 데이터로 같은 장소에서 4번씩 12월, 1월, 2월, 3월 이렇게 측정되었으며 지방에 따라 Fat weight와 Lean dry weight로 나뉜 데이터이다.
- 이 데이터를 사용하여 그룹별로 지방 함량의 평균이 같은지 알아보기 위해 다변량 분산분석을 시행해보고자 한다.
- 본 검정의 가설은 다음과 같다.
- 우선 반복측정자료분석에서의 가정 (다변량적 접근)을 살펴보겠다.
- 종속변수들의 상관성
- 다변량 정규성 (multivariate normality)
- 종속변수들은 다변량 정규분포를 따른다.
- 종속변수들의 선형조합도 정규분포를 따른다.
- 종속변수들은 다변량 정규분포를 따른다.
- 분산-공분산 행렬의 동질성 (homogeneity)
- MANOVA는 그룹내 분산-공분산 행렬이 동일하다고 가정한다.
- 분산-공분산 행렬의 동질성은 Box의 M-test를 이용하여 확인한다.
- MANOVA는 그룹내 분산-공분산 행렬이 동일하다고 가정한다.
- 선형성 (linearity): 종속변수들과 독립변수들 간 선형성
- 관측 벡터의 독립성 (independence): 각 개체들은 서로 독립
- 임의 표본 (random sampling)
- 종속변수들의 상관성
- 다음은은 MANOVA에 대한 검정통계량을 살펴보겠다.
- Wilks’ lambda (윌크스의 람다)
- 0과 1사이의 값으로 산출되며, 처리효과가 있을수록 값이 작아진다.
- 만약 그룹 간 효과와 관련된 B(between)가 커지면 윌크스의 람다 값은 0에 가까워지고 만약 작아지면 1에 가까워진다.
\(\Lambda=\frac{\mid W \mid }{\mid B+W \mid}=\frac{\mid W \mid}{\mid T \mid}=\prod_{i=1}^{q}\frac{1}{1+\lambda_i}\)
\(where \ W \ and \ T \ are \ determinants \ of \ the \ within \ and \ total \ sum \ of \ squares \ and \ cross-product \ matrices\)
- Phillai’s trace (필라이 대각합)
- 양수의 값을 가지며, 처리효과가 클수록 값이 커진다.
\(trace[B(B+W)^{-1}]=\sum_{i=1}^{q}\frac{\lambda_i}{1+\lambda_i}\)
- 양수의 값을 가지며, 처리효과가 클수록 값이 커진다.
- Lawley-Hotelling trace (로리-호텔링의 대각합)
- 양수의 값을 가지며, 효과가 있으면 값이 커진다.
\(trace[BW^{-1}]=\sum_{i=1}^{q}\lambda_i\)
- 양수의 값을 가지며, 효과가 있으면 값이 커진다.
- Roy’s maximum root (로이의 최대근)
- 양수의 값을 가지며, 효과가 있을수록 값이 커진다.
\(max(\lambda_i)\)
- 양수의 값을 가지며, 효과가 있을수록 값이 커진다.
- Wilks’ lambda (윌크스의 람다)
- 데이터에 이상치가 존재하는지 확인하도록 하겠다.
library("mvoutlier")
aq.plot(na.omit(ex16[,2:3]),alpha=0.05)
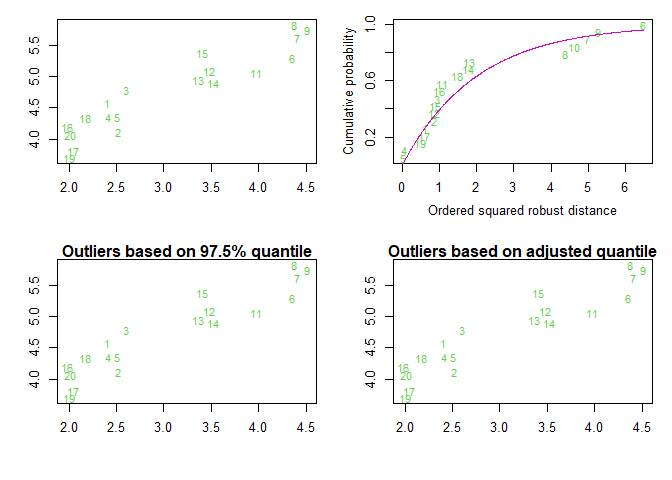
## $outliers
## 1 2 3 4 5 6 7 8 9 11 12 13 14
## FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## 15 16 17 18 19 20
## FALSE FALSE FALSE FALSE FALSE FALSE
- 이상치가 없는 것으로 확인되며, 그룹별로 정규성을 따르는지 확인하도록 하겠다.
다변량 정규성 검정
library("mvnormtest") # mshapiro.test
library("broom") # tidy
mshapiro.test(t(ex16[1:5,2:3])) %>% tidy %>%
kable(,caption = "Normality test (December)") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| statistic | p.value | method |
|---|---|---|
| 0.79349 | 0.0716398 | Shapiro-Wilk normality test |
mshapiro.test(t(na.omit(ex16[6:10,2:3]))) %>% tidy %>%
kable(,caption = "Normality test (January)") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| statistic | p.value | method |
|---|---|---|
| 0.8272233 | 0.1606765 | Shapiro-Wilk normality test |
mshapiro.test(t(ex16[11:15,2:3])) %>% tidy %>%
kable(,caption = "Normality test (February)") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| statistic | p.value | method |
|---|---|---|
| 0.7764365 | 0.0513539 | Shapiro-Wilk normality test |
mshapiro.test(t(ex16[16:20,2:3])) %>% tidy %>%
kable(,caption = "Normality test (March)") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| statistic | p.value | method |
|---|---|---|
| 0.7946881 | 0.0732947 | Shapiro-Wilk normality test |
- 정규성 검정 결과 모든 p-value는 유의수준 0.05보다 크므로 정규성을 따른다는 귀무가설을 기각할 충한 근거가 없다.
그러므로 해당 데이터는 그룹별로 정규성을 따른다고 하겠다. - 다음으로는 월 별로 공분산 행렬을 계산하여 보고, 공분산 행렬이 동질적인지 Box’s M test를 수행하도록 하겠다.
분산-공분산행렬 동질성 검정
ex16_na <- na.omit(ex16)
covm <- by(ex16_na[,2:3], ex16_na$month, cov)
covm$December %>%
kable(caption = "Covariance Matrices by December") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| fat | lean | |
|---|---|---|
| fat | 0.00673 | 0.00662 |
| lean | 0.00662 | 0.06568 |
covm$January %>%
kable(caption = "Covariance Matrices by January") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| fat | lean | |
|---|---|---|
| fat | 0.004825 | 0.0086250 |
| lean | 0.008625 | 0.0544917 |
covm$February %>%
kable(caption = "Covariance Matrices by February") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| fat | lean | |
|---|---|---|
| fat | 0.061600 | -0.006375 |
| lean | -0.006375 | 0.035030 |
covm$March %>%
kable(caption = "Covariance Matrices by March") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| fat | lean | |
|---|---|---|
| fat | 0.00563 | 0.01001 |
| lean | 0.01001 | 0.06827 |
library("biotools") # boxM
boxM(ex16_na[,2:3], ex16_na$month) %>% tidy %>%
rename("Chi-sq statistic"="statistic","df"="parameter") %>%
kable(caption = "Homogeneity of Covariance Matrices") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Chi-sq statistic | p.value | df | method |
|---|---|---|---|
| 9.559461 | 0.3873108 | 9 | Box’s M-test for Homogeneity of Covariance Matrices |
- Box’s M test의 결과 p-value=0.387로써 유의수준 5%하에 귀무가설을 기각하지 못한다.
- 따라서 그룹간 공분산 행렬이 동일하지 않다는 충분한 근거가 없으며, 구형성을 만족한다고 할 수 있다.
- 이제 위에서 소개했던 MANOVA에 대한 검정통계량을 적용하여 보겠다.
anov <- manova(cbind(fat,lean)~month, data=ex16_na)
tidy(anov, test = "Wilks") %>%
rename("Wilk's Lambda"="wilks","F value"="statistic") %>%
kable(caption = "MANOVA using Wilk's Lambda",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | df | Wilk’s Lambda | F value | num.df | den.df | p.value |
|---|---|---|---|---|---|---|
| month | 3 | 0.0177815 | 30.32966 | 6 | 28 | 0 |
| Residuals | 15 | NA | NA | NA | NA | NA |
tidy(anov) %>% rename("Pillai's trace"="pillai","F value"="statistic") %>%
kable(caption = "MANOVA using Pillai's trace",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | df | Pillai’s trace | F value | num.df | den.df | p.value |
|---|---|---|---|---|---|---|
| month | 3 | 1.02229 | 5.227982 | 6 | 30 | 0.0008709 |
| Residuals | 15 | NA | NA | NA | NA | NA |
tidy(anov, test = "Hotelling") %>% rename("Hotelling-Lawley trace"="hl","F value"="statistic") %>%
kable(caption = "MANOVA using Hotelling-Lawley trace",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | df | Hotelling-Lawley trace | F value | num.df | den.df | p.value |
|---|---|---|---|---|---|---|
| month | 3 | 52.98465 | 114.8001 | 6 | 26 | 0 |
| Residuals | 15 | NA | NA | NA | NA | NA |
tidy(anov, test = "Roy") %>% rename("Roy's maximum root"="roy","F value"="statistic") %>%
kable(caption = "MANOVA using Roy's maximum root",booktabs = TRUE, valign = 't') %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| term | df | Roy’s maximum root | F value | num.df | den.df | p.value |
|---|---|---|---|---|---|---|
| month | 3 | 52.94209 | 264.7104 | 3 | 15 | 0 |
| Residuals | 15 | NA | NA | NA | NA | NA |
- 4가지 방법 모두 p-value가 유의수준 0.05보다 작은 값을 가지는 것을 볼 수 있다.
- 따라서 월별 모든 참새들의 fat weight와 lean dry weight가 다르다고 할 수 있다.
- 다변량 분산분석의 사후분석을 하기 위해 판별함수 분석을 수행하여 종속변수들이 그룹을 어떻게 분리하는지 파악하도록 하겠다.
사후분석
dfa <- lda(month~fat+lean,data=ex16_na)
dfa
## Call:
## lda(month ~ fat + lean, data = ex16_na)
##
## Prior probabilities of groups:
## December February January March
## 0.2631579 0.2631579 0.2105263 0.2631579
##
## Group means:
## fat lean
## December 2.4940 4.4360
## February 3.5500 5.0740
## January 4.4125 5.6225
## March 2.0440 4.0180
##
## Coefficients of linear discriminants:
## LD1 LD2
## fat 6.461863 2.72530
## lean 1.127769 -4.11224
##
## Proportion of trace:
## LD1 LD2
## 0.9992 0.0008
- Prior probabilities of groups를 보면 4개의 월별 모두 표본 크기가 같으므로 사전확률도 0.2631579로 같다.
- 첫번쨰 판별함수 LD1의 열을 보면 fat weight 와 lean dry weight의 효과를 같은 방향으로 구분한다.
- 두번쨰 판별함수 LD2의 열을 보면 fat weight 와 lean dry weight의 효과를 반대 방향으로 구분한다.
- Proportion of trace를 보면 LD1은 전체 변동의 99.92%를, LD2는 전체 변동의 0.08%를 설명한다.
- 마지막으로 판별점수를 가지고 산점도를 그려 시각화하여 보겠다.
lda_df <- data.frame(
month = ex16_na[, "month"],
lda = predict(dfa)$x
)
library("ggplot2")
ggplot(lda_df) +
geom_point(aes(x = lda.LD1, y = lda.LD2, color = month), size = 4) +
theme_classic()
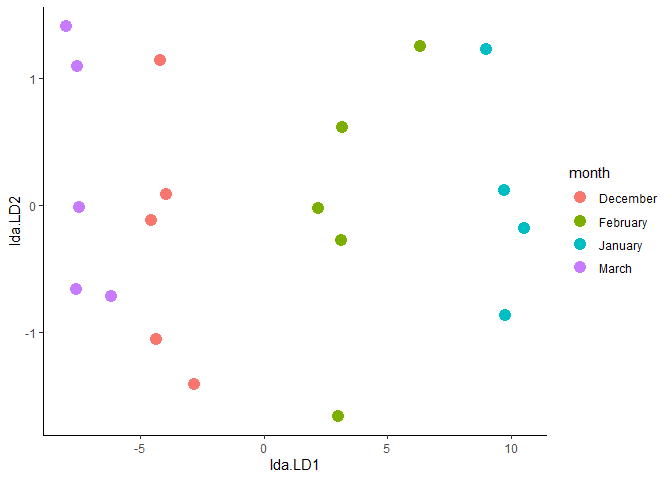
- 그래프를 보면 December과 March일 때의 판별점수가 February와 January와 확실히 significant하게 다름을 볼 수 있다.
- December일 때와 March일 때가 매우 비슷하며 January 그룹에서 그룹별 지방함량의 평균이 같다는 귀무가설을 기각할 충분한 근거가 있는 효과 크기를 가지고 있다고 볼 수 있다.
SAS 프로그램 결과
SAS 접기/펼치기 버튼
16장
EXAMPLE 16.1
LIBNAME ex 'C:\Biostat';
RUN;
PROC IMPORT DBMS=sav
DATAFILE='C:\Biostat\Exam 16.1.sav'
OUT=ex.ex16_1 REPLACE;
RUN;
DATA ex.ex16_1x;
INPUT month $ w $ @@;
CARDS;
12 F 12 L
1 F 1 L
2 F 2 L
3 F 3 L
;
RUN;
PROC TRANSPOSE DATA=ex.ex16_1 OUT=ex.ex16_1a;
RUN;
DATA ex.ex16_1b;
MERGE ex.ex16_1x ex.ex16_1a;
RUN;
PROC SORT DATA=ex.ex16_1b;
BY month;
RUN;
PROC TRANSPOSE DATA=ex.ex16_1b OUT=ex.ex16_1c (drop=_NAME_);
BY month;
ID w;
VAR col1-col5;
RUN;
/*분산-공분산행렬 동질성 검정*/
PROC DISCRIM DATA=ex.ex16_1c POOL=test WCOV;
CLASS month;
VAR F L;
RUN;
The DISCRIM Procedure
| Total Sample Size | 19 | DF Total | 18 |
|---|---|---|---|
| Variables | 2 | DF Within Classes | 15 |
| Classes | 4 | DF Between Classes | 3 |
| Number of Observations Read | 20 |
|---|---|
| Number of Observations Used | 19 |
| Class Level Information | |||||
|---|---|---|---|---|---|
| month | Variable Name | Frequency | Weight | Proportion | Prior Probability |
| 1 | 1 | 4 | 4.0000 | 0.210526 | 0.250000 |
| 12 | 12 | 5 | 5.0000 | 0.263158 | 0.250000 |
| 2 | 2 | 5 | 5.0000 | 0.263158 | 0.250000 |
| 3 | 3 | 5 | 5.0000 | 0.263158 | 0.250000 |
The DISCRIM Procedure
Within-Class Covariance Matrices
| month = 1, DF = 3 | ||
|---|---|---|
| Variable | F | L |
| F | 0.0048250000 | 0.0086250000 |
| L | 0.0086250000 | 0.0544916667 |
| month = 12, DF = 4 | ||
|---|---|---|
| Variable | F | L |
| F | 0.0067300000 | 0.0066200000 |
| L | 0.0066200000 | 0.0656800000 |
| month = 2, DF = 4 | ||
|---|---|---|
| Variable | F | L |
| F | 0.0616000000 | -.0063750000 |
| L | -.0063750000 | 0.0350300000 |
| month = 3, DF = 4 | ||
|---|---|---|
| Variable | F | L |
| F | 0.0056300000 | 0.0100100000 |
| L | 0.0100100000 | 0.0682700000 |
| Within Covariance Matrix Information | ||
|---|---|---|
| month | Covariance Matrix Rank | Natural Log of the Determinant of the Covariance Matrix |
| 1 | 2 | -8.57624 |
| 12 | 2 | -7.82855 |
| 2 | 2 | -6.15766 |
| 3 | 2 | -8.16597 |
| Pooled | 2 | -6.77867 |
The DISCRIM Procedure
Test of Homogeneity of Within Covariance Matrices
| Chi-Square | DF | Pr > ChiSq |
|---|---|---|
| 9.559461 | 9 | 0.3873 |
Since the Chi-Square value is not significant at the 0.1 level, a pooled covariance matrix will be used in the discriminant function.
Reference: Morrison, D.F. (1976) Multivariate Statistical Methods p252.
The DISCRIM Procedure
| Generalized Squared Distance to month | ||||
|---|---|---|---|---|
| From month | 1 | 12 | 2 | 3 |
| 1 | 0 | 188.77724 | 38.34912 | 292.92415 |
| 12 | 188.77724 | 0 | 56.96520 | 11.66189 |
| 2 | 38.34912 | 56.96520 | 0 | 119.35753 |
| 3 | 292.92415 | 11.66189 | 119.35753 | 0 |
| Linear Discriminant Function for month | ||||
|---|---|---|---|---|
| Variable | 1 | 12 | 2 | 3 |
| Constant | -668.95259 | -288.49311 | -473.36844 | -217.32186 |
| F | 194.98174 | 105.27489 | 154.71136 | 84.78096 |
| L | 84.93522 | 70.88157 | 78.34283 | 65.04515 |
The DISCRIM Procedure
Classification Summary for Calibration Data: EX.EX16_1C
Resubstitution Summary using Linear Discriminant Function
| Number of Observations and Percent Classified into month | |||||
|---|---|---|---|---|---|
| From month | 1 | 12 | 2 | 3 | Total |
| 1 | 4 100.00 | 0 0.00 | 0 0.00 | 0 0.00 | 4 100.00 |
| 12 | 0 0.00 | 5 100.00 | 0 0.00 | 0 0.00 | 5 100.00 |
| 2 | 0 0.00 | 0 0.00 | 5 100.00 | 0 0.00 | 5 100.00 |
| 3 | 0 0.00 | 0 0.00 | 0 0.00 | 5 100.00 | 5 100.00 |
| Total | 4 21.05 | 5 26.32 | 5 26.32 | 5 26.32 | 19 100.00 |
| Priors | 0.25 | 0.25 | 0.25 | 0.25 | |
| Error Count Estimates for month | |||||
|---|---|---|---|---|---|
| 1 | 12 | 2 | 3 | Total | |
| Rate | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 |
| Priors | 0.2500 | 0.2500 | 0.2500 | 0.2500 | |
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex16_1c;
CLASS month;
MODEL F L=month;
MANOVA H=month;
ODS OUTPUT OverallANOVA=OverallANOVA MultStat=MultStat;
RUN;
QUIT;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=OverallANOVA;
RUN;
PROC PRINT DATA=MultStat;
RUN;
| OBS | Dependent | Source | DF | SS | MS | FValue | ProbF |
|---|---|---|---|---|---|---|---|
| 1 | F | Model | 3 | 15.28045342 | 5.09348447 | 246.21 | <.0001 |
| 2 | F | Error | 15 | 0.31031500 | 0.02068767 | _ | _ |
| 3 | F | Corrected Total | 18 | 15.59076842 | _ | _ | _ |
| 4 | L | Model | 3 | 6.74124711 | 2.24708237 | 40.16 | <.0001 |
| 5 | L | Error | 15 | 0.83939500 | 0.05595967 | _ | _ |
| 6 | L | Corrected Total | 18 | 7.58064211 | _ | _ | _ |
| OBS | Hypothesis | Error | Statistic | Value | FValue | NumDF | DenDF | ProbF | PValue |
|---|---|---|---|---|---|---|---|---|---|
| 1 | month | Error SSCP Matrix | Wilks' Lambda | 0.01778151 | 30.33 | 6 | 28 | <.0001 | . |
| 2 | month | Error SSCP Matrix | Pillai's Trace | 1.02229007 | 5.23 | 6 | 30 | 0.0009 | . |
| 3 | month | Error SSCP Matrix | Hotelling-Lawley Trace | 52.98465188 | 120.10 | 6 | 17 | <.0001 | . |
| 4 | month | Error SSCP Matrix | Roy's Greatest Root | 52.94208549 | 264.71 | 3 | 15 | <.0001 | . |
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
