[의학통계방법론] Ch10. Multisample Hypotheses and the Analysis of Variance
Multisample Hypotheses and
the Analysis of Variance
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("readxl")
library("dplyr")
library("kableExtra")
library("randtests")
library("lmtest")
library("car")
library("lawstat")
library("onewaytests")
library("pwr2")
library("pgirmess")
library("DescTools")
library("RVAideMemoire")
엑셀파일불러오기
#모든 시트를 하나의 리스트로 불러오는 함수
read_excel_allsheets <- function(file, tibble = FALSE) {
sheets <- readxl::excel_sheets(file)
x <- lapply(sheets, function(X) readxl::read_excel(file, sheet = X, na = "."))
if(!tibble) x <- lapply(x, as.data.frame)
names(x) <- sheets
x
}
10장
10장 연습문제 불러오기
#data_chap10에 연습문제 10장 모든 문제 저장
data_chap10 <- read_excel_allsheets("data_chap10.xls")
#연습문제 각각 데이터 생성
for (x in 1:length(data_chap10)){
assign(paste0('ex10_',c(1:3,10:13))[x],data_chap10[x])
}
#연습문제 데이터 형식을 리스트에서 데이터프레임으로 변환
for (x in 1:length(data_chap10)){
assign(paste0('ex10_',c(1:3,10:13))[x],data.frame(data_chap10[x]))
}
EXAMPLE 10.1
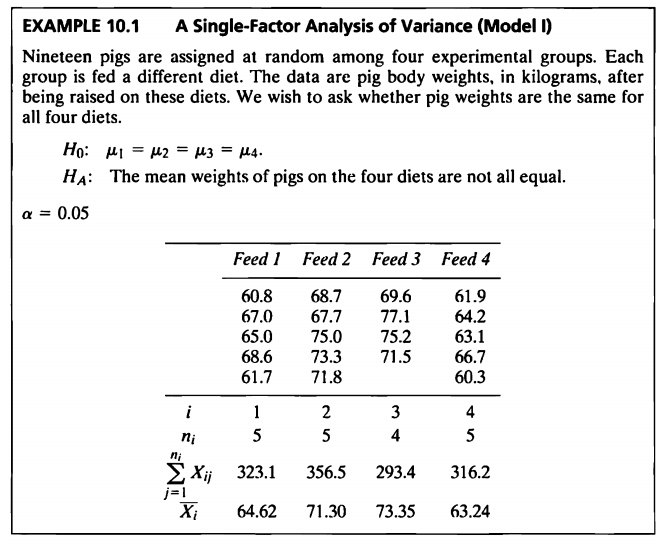
#데이터셋
ex10_1%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_1.id | exam10_1.diet | exam10_1.weight |
|---|---|---|
| 1 | 1 | 60.8 |
| 2 | 1 | 67.0 |
| 3 | 1 | 65.0 |
| 4 | 1 | 68.6 |
| 5 | 1 | 61.7 |
| 6 | 2 | 68.7 |
| 7 | 2 | 67.7 |
| 8 | 2 | 75.0 |
| 9 | 2 | 73.3 |
| 10 | 2 | 71.8 |
| 11 | 3 | 69.6 |
| 12 | 3 | 77.1 |
| 13 | 3 | 75.2 |
| 14 | 3 | 71.5 |
| 15 | 3 | NA |
| 16 | 4 | 61.9 |
| 17 | 4 | 64.2 |
| 18 | 4 | 63.1 |
| 19 | 4 | 66.7 |
| 20 | 4 | 60.3 |
데이터: 19마리 돼지를 4개 그룹으로 무작위로 나누어 서로 다른 다이어트 처리 후 중량(kg) 측정
- 이 문제의 데이터는 돼지의 체중(weight) 증가에 가장 효율적인 사료(feed)를 찾기 위한 실험에서 얻어진 측정값들이다.
- 19마리의 돼지가 각 처리별로 랜덤배정되었다.
- 사료의 종류에 따라 나뉘어진 네 집단 간 무게의 모평균에 차이가 있는지를 검정할 것이다.
- 단일 요인 분산 분석 ANOVA는 완전히 랜덤화 된 실험 설계 방법이다.
- “completely randomized design”이라고도 한다.
- 일반적으로 데이터에서 그룹의 통계적 비교는 각 그룹의 데이터 수가 동일한 경우(균형적인 상황)에 가장 적합하다.
- 균형적일 때 검정의 검정력이 높아진다.
- 하지만 10.1의 데이터의 경우 4개의 그룹 각각에 5마리의 실험동물이 있지만,
Feed 3에 속한 한 마리의 동물의 몸무게는 어떤 적절한 이유로 분석에 사용되지 않았다.
아니면 병이 났거나 임신한 것으로 밝혀졌을 수도 있다. - 분산 분석에서는 실험 요인을 제외한 모든 면에서 가능한 유사해야 한다.
(즉, 동물은 동일한 품종, 성별, 연령이어야 하고, 같은 온도로 유지되어야 한다.)
- 분석을 위한 통계량
ex10_1 %>%
group_by(exam10_1.diet) %>%
summarize(mean=mean(exam10_1.weight,na.rm=T),sum=sum(exam10_1.weight,na.rm=T))%>%
kbl(caption = "Dataset") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_1.diet | mean | sum |
|---|---|---|
| 1 | 64.62 | 323.1 |
| 2 | 71.30 | 356.5 |
| 3 | 73.35 | 293.4 |
| 4 | 63.24 | 316.2 |
EXAMPLE 10.1a
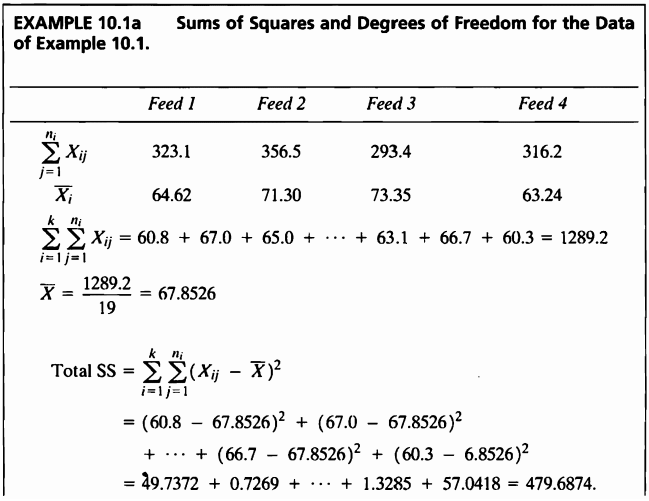

- 분석을 위해 결측치 제거해준 뒤 자유도와 제곱합들을 구하도록 한다.
ex10_1[15,3]=NA
ex10_1<-na.omit(ex10_1)
ex10_1a <- function(x){
sum <- ex10_1 %>% summarize(sum=sum(exam10_1.weight,na.rm=T))
mean <- ex10_1 %>% summarize(mean=mean(exam10_1.weight,na.rm=T))
total_df<-length(x)-1
groups_df<-length(unique(ex10_1$exam10_1.diet))-1
error_df<-length(x)-length(unique(ex10_1$exam10_1.diet))
y <- rep(0,4)
for (i in 1:4){
y[i]=length(ex10_1[ex10_1$exam10_1.diet==i,3])*((mean(ex10_1[ex10_1$exam10_1.diet==i,3])-mean(x))^2)
}
SSB=sum(y)
SST=sum((x-mean(x))^2)
SSE=SST-SSB
table <- data.frame(total_DF=total_df,groups_DF=groups_df,error_DF=error_df,Total_SS=SST,Group_SS=SSB,Error_SS=SSE)
return(table)
}
ex10_1a(ex10_1$exam10_1.weight)%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| total_DF | groups_DF | error_DF | Total_SS | Group_SS | Error_SS |
|---|---|---|---|---|---|
| 18 | 3 | 15 | 479.6874 | 338.9374 | 140.75 |
EXAMPLE 10.1b

- “Machine Formulas”라는 새로운 방식을 사용한 결과는 다음과 같다.
ex10_1b <- function(){
sum <- ex10_1 %>% summarize(sum=sum(exam10_1.weight,na.rm=T))
sum_s <- ex10_1 %>% summarize(sum_s=sum((exam10_1.weight)^2,na.rm=T))
c <- sum^2/length(ex10_1$exam10_1.weight)
y <- rep(0,4)
for (i in 1:4){
y[i]=(sum(ex10_1[ex10_1$exam10_1.diet==i,3])^2)/(length(ex10_1[ex10_1$exam10_1.diet==i,3]))
}
ss <- sum(y)
tss <- round(sum_s-c,4)
gss <- (ss-c)
ess <- (round(sum_s-c,4)-(ss-c))
table <- data.frame(tss,gss,ess)
names(table) <- c("Total_SS","Group_SS","Error_SS")
return(table)
}
ex10_1b()%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| Total_SS | Group_SS | Error_SS |
|---|---|---|
| 479.6874 | 338.9374 | 140.75 |
- “Machine Formulas” 방법을 사용한 결과와 10.1a에서 구한 결과가 같은 것을 확인할 수 있다.
- 예제 10.1b와 같이 간단한 계산기를 사용할 때 이와 같은 기계식은 매우 편리할 수 있지만, 통계적 계산을 컴퓨터에 의해 수행하는 경우에는 덜 중요하다.
EXAMPLE 10.1c
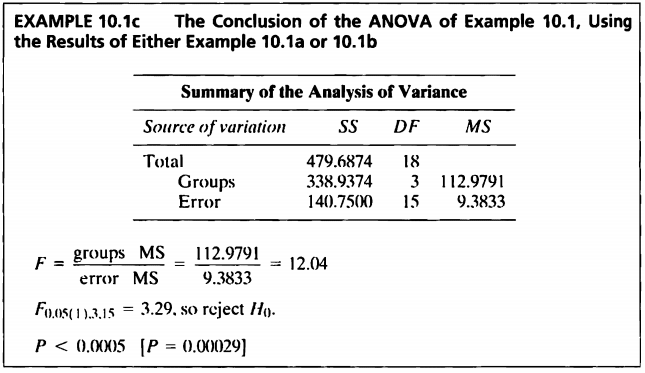
- 10.1a와 10.1b에서 구한 값들을 토대로 ANOVA를 진행하도록 한다.
- 일원분산분석을 진행하기 전에 정규성과 등분산성, 독립성을 만족하는지 부터 확인하도록 한다.
Shapiro-Wilk Test로 네 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_1$exam10_1.weight,ex10_1$exam10_1.diet==1));shapiro.test(subset(ex10_1$exam10_1.weight,ex10_1$exam10_1.diet==2));shapiro.test(subset(ex10_1$exam10_1.weight,ex10_1$exam10_1.diet==3));shapiro.test(subset(ex10_1$exam10_1.weight,ex10_1$exam10_1.diet==4))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_1$exam10_1.weight, ex10_1$exam10_1.diet == 1)
## W = 0.93315, p-value = 0.618
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_1$exam10_1.weight, ex10_1$exam10_1.diet == 2)
## W = 0.94389, p-value = 0.6936
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_1$exam10_1.weight, ex10_1$exam10_1.diet == 3)
## W = 0.95202, p-value = 0.7287
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_1$exam10_1.weight, ex10_1$exam10_1.diet == 4)
## W = 0.99027, p-value = 0.9806
- 네 그룹 모두 정규성 검정 결과 p-value가 0.05보다 매우 크므로 네 그룹 모두 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 따라서 정규성을 만족한다는 사실을 바탕으로 그룹별 등분산성 검정을 진행한다.
Bartlett’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
bartlett.test(ex10_1$exam10_1.weight~ex10_1$exam10_1.diet)
##
## Bartlett test of homogeneity of variances
##
## data: ex10_1$exam10_1.weight by ex10_1$exam10_1.diet
## Bartlett's K-squared = 0.47515, df = 3, p-value = 0.9243
- Bartlett’s Test의 유의확률은 p-value = 0.9243으로 유의수준 0.05보다 매우 크므로 등분산성이 아니라는 대립가설을 채택할 근거가 없다.
- Runs Test와 Durbin-Watson test을 통하여 잔차의 자기상관성을 확인하여 보겠다.
Runs Test
anova_ex10_1 <- aov(ex10_1$exam10_1.weight~as.factor(ex10_1$exam10_1.diet))
library(randtests)
runs.test(anova_ex10_1$residuals, alternative = "two.sided", threshold = 0.0, plot = TRUE)
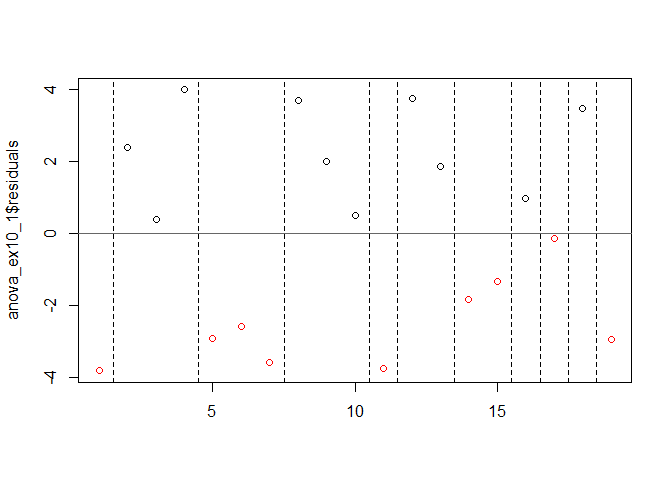
##
## Runs Test - Two sided
##
## data: anova_ex10_1$residuals
## Standardized Runs Statistic = 0.24922, p-value = 0.8032
- 잔차 그림을 확인하여 보면 뚜렷한 패턴이 보이지 않았다.
- Runs Test의 경우 양의 계열상관을 검정하기 위해서는 하한임계치를 사용하고,
음의 계열상관을 검정하기 위해서는 상한 임계치를 사용하며, 계열상관의 존재 여부를 위해서는 양쪽 임계치 모두를 사용한다. - 양측 검정을 시행하였으며, p-value = 0.8032이므로 귀무가설 “연속적인 관측값이 임의적이다.”를 기각하지 못한다.
Durbin-Watson test
library(lmtest)
dwtest(anova_ex10_1, alternative = "greater")
##
## Durbin-Watson test
##
## data: anova_ex10_1
## DW = 2.2368, p-value = 0.4203
## alternative hypothesis: true autocorrelation is greater than 0
- Durbin-Watson test의 경우 DW = 2.2368으로 4보다 2와 가깝게 나왔으며, 2보다 크므로 alternative = “greater” 주었고 p-value = 0.4203으로 귀무가설 “자기상관이 0 이다.”를 기각하지 못한다.
- 자기상관성이 없다고 독립인 건 아니지만 독립성 가정이 만족된다고 가정할 수 있을 것 같다.
- Shapiro-Wilk test와 Bartlett’s Test으로 정규성과 등분산성을 확인하였으므로 ANOVA 검정을 진행한다.
class(ex10_1$exam10_1.diet)
## [1] "numeric"
- diet의 class가 numeric이므로 factor로 변경해서 ANOVA 검정을 실시한다.
summary(aov(ex10_1$exam10_1.weight~as.factor(ex10_1$exam10_1.diet)))
## Df Sum Sq Mean Sq F value Pr(>F)
## as.factor(ex10_1$exam10_1.diet) 3 338.9 112.98 12.04 0.000283 ***
## Residuals 15 140.8 9.38
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
- p-value가 0.000283으로 유의수준 0.05보다 매우 작기 때문에 귀무가설을 기각할 수 있고 따라서 유의수준 5%하에 식단에 따른 돼지의 몸무게 모평균 차이가 있다고 말할 수 있다.
EXAMPLE 10.2

#데이터셋
ex10_2
## exam10_2.id exam10_2.technician exam10_2.phosphorus
## 1 1 1 34
## 2 2 1 36
## 3 3 1 34
## 4 4 1 35
## 5 5 1 34
## 6 6 2 37
## 7 7 2 36
## 8 8 2 35
## 9 9 2 37
## 10 10 2 37
## 11 11 3 34
## 12 12 3 37
## 13 13 3 35
## 14 14 3 37
## 15 15 3 36
## 16 16 4 36
## 17 17 4 34
## 18 18 4 37
## 19 19 4 34
## 20 20 4 35
- 실험실에서 건초의 인 함량을 측정하는 기술을 사용하는데
“인 측정값이 분석을 수행하는 기술자마다 다릅니까?”라는 질문이 발생한다. - 이 질문에 답하기 위해 무작위로 선택된 네 명의 기술자 각각에게 동일한 건초 배치에서 5개의 샘플이 주어졌고, 20개의 인 측정 결과(건초 mg/g)가 표시된다.
- Example 10.2는 랜덤 효과 모형에 대한 분산 분석을 보여준다.
- Example 10.2의 심각한 결과는 기본 가정으로부터 이탈하는 경우이다.
- 다행히도 많은 상황에서 분산 분석은 강력한 테스트이며, 이는 제 1종오류, 제 2종오류 확률이 항상 test 가정 위반에 의해 심각하게 변경되는 것은 아니라는 것을 의미한다.
- two-sample t-test과 마찬가지로 비정규성의 부작용은 정규성에서 벗어날수록 더 크지만 표본 크기가 같을 경우 효과는 상대적으로 작다.
- 본 검정의 가설은 다음과 같다.
- F 통계량을 구하는 공식은 다음과 같다.
- 이를 분산분석을 통해 알아보기 위해 정규성과 등분산성을 만족하는지 확인하도록 한다.
Shapiro-Wilk Test로 네 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_2$exam10_2.phosphorus,ex10_2$exam10_2.technician==1));shapiro.test(subset(ex10_2$exam10_2.phosphorus,ex10_2$exam10_2.technician==2));shapiro.test(subset(ex10_2$exam10_2.phosphorus,ex10_2$exam10_2.technician==3));shapiro.test(subset(ex10_2$exam10_2.phosphorus,ex10_2$exam10_2.technician==4))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_2$exam10_2.phosphorus, ex10_2$exam10_2.technician == 1)
## W = 0.77091, p-value = 0.04595
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_2$exam10_2.phosphorus, ex10_2$exam10_2.technician == 2)
## W = 0.77091, p-value = 0.04595
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_2$exam10_2.phosphorus, ex10_2$exam10_2.technician == 3)
## W = 0.90202, p-value = 0.4211
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_2$exam10_2.phosphorus, ex10_2$exam10_2.technician == 4)
## W = 0.90202, p-value = 0.4211
- Shapiro-Wilk Test로 1,2 그룹은 모두 정규성 검정한 결과 p-value가 0.05보다 작으므로 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 있고, 3,4 그룹은 모두 정규성 검정한 결과 p-value가 0.05보다 매우 크므로 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 등분산성 검정으로 Bartlett’s Test가 아닌 normal상태가 아닌것에 대해 덜 민감한 Levene’s Test를 진행하여 보자.
class(ex10_2$exam10_2.technician)
## [1] "numeric"
- technician의 class가 numeric이므로 factor로 변경해서 등분산성 검정을 실시한다.
- R에서 car, lawstat 패키지에서 Levene’s Test를 제공하여 준다.
Levene’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
library(car)
leveneTest(ex10_2$exam10_2.phosphorus~as.factor(ex10_2$exam10_2.technician),center=mean)
## Levene's Test for Homogeneity of Variance (center = mean)
## Df F value Pr(>F)
## group 3 0.6827 0.5755
## 16
library(lawstat)
levene.test(ex10_2$exam10_2.phosphorus,as.factor(ex10_2$exam10_2.technician), location="mean")
##
## Classical Levene's test based on the absolute deviations from the mean
## ( none not applied because the location is not set to median )
##
## data: ex10_2$exam10_2.phosphorus
## Test Statistic = 0.68267, p-value = 0.5755
- p-value = 0.5755으로 유의수준 0.05보다 매우 크므로 등분산성이 아니라는 대립가설을 채택할 근거가 없다.
- Runs Test와 Durbin-Watson test을 통하여 잔차의 자기상관성을 확인하여 보겠다.
Runs Test
anova_ex10_2 <- aov(ex10_2$exam10_2.phosphorus~as.factor(ex10_2$exam10_2.technician))
runs.test(anova_ex10_2$residuals, alternative = "two.sided", threshold = 0, plot = TRUE)
##
## Runs Test - Two sided
##
## data: anova_ex10_2$residuals
## Standardized Runs Statistic = 1.8379, p-value = 0.06608
- p-value = 0.06608로써 만족스럽게 귀무가설을 기각하지 못하였다.
Durbin-Watson test
dwtest(anova_ex10_2, alternative = "greater")
##
## Durbin-Watson test
##
## data: anova_ex10_2
## DW = 3.228, p-value = 0.994
## alternative hypothesis: true autocorrelation is greater than 0
- Durbin-Watson test의 경우 DW = 3.228로써 4에 가까운 값이 나왔으며, 2보다 크므로 alternative = “greater” 주었고 p-value = 0.994로 귀무가설 “자기상관이 0 이다.”를 기각하지 못한다.
- 자기상관성이 없다고 독립인 건 아니지만 독립성 가정이 만족된다고 가정할 수 있을 것 같다.
- 표본이 정규분포를 따르지 않지만 유사한 분포를 가지고 있고 분산도 유사하다면 Kruskal-Wallis Test가 적절하다.
kruskal.test(ex10_2$exam10_2.phosphorus~as.factor(ex10_2$exam10_2.technician))
##
## Kruskal-Wallis rank sum test
##
## data: ex10_2$exam10_2.phosphorus by as.factor(ex10_2$exam10_2.technician)
## Kruskal-Wallis chi-squared = 6.0065, df = 3, p-value = 0.1113
- Kruskal-Wallis Test 결과 p-value가 0.1113으로 유의수준 0.05 보다 크기 때문에 귀무가설을 기각할 수 없고 따라서 유의수준 5%하에 인 함량의 모평균이 기술자마다 다르다고 말할만한 충분한 근거가 없다고 말할 수 있다.
EXAMPLE 10.3


#데이터셋
ex10_3
## exam10_3.id exam10_3.variety exam10_3.potassium
## 1 1 1 27.9
## 2 2 1 27.0
## 3 3 1 26.0
## 4 4 1 26.5
## 5 5 1 27.0
## 6 6 1 27.5
## 7 7 2 24.2
## 8 8 2 24.7
## 9 9 2 25.6
## 10 10 2 26.0
## 11 11 2 27.4
## 12 12 2 26.1
## 13 13 3 29.1
## 14 14 3 27.7
## 15 15 3 29.9
## 16 16 3 30.7
## 17 17 3 28.8
## 18 18 3 31.1
- 포타슘 함량(식물 조직 100mg당 포타슘의 mg)은 세 가지 종류의 밀 각각 6개의 모종에서 측정되었다.
- 본 검정의 가설은 다음과 같다.
Shapiro-Wilk Test로 네 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_3$exam10_3.potassium,ex10_3$exam10_3.variety==1));shapiro.test(subset(ex10_3$exam10_3.potassium,ex10_3$exam10_3.variety==2));shapiro.test(subset(ex10_3$exam10_3.potassium,ex10_3$exam10_3.variety==3))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_3$exam10_3.potassium, ex10_3$exam10_3.variety == 1)
## W = 0.9778, p-value = 0.9401
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_3$exam10_3.potassium, ex10_3$exam10_3.variety == 2)
## W = 0.96651, p-value = 0.8682
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_3$exam10_3.potassium, ex10_3$exam10_3.variety == 3)
## W = 0.96881, p-value = 0.8844
- Shapiro-Wilk test 결과 p-value가 0.05보다 매우 크므로 세 그룹 모두 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 따라서 정규성을 만족한다는 사실을 바탕으로 그룹별 등분산성 검정을 진행한다.
class(ex10_3$exam10_3.variety)
## [1] "numeric"
- variety의 class가 numeric이므로 factor로 변경해서 등분산성 검정을 실시한다.
Bartlett’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
bartlett.test(ex10_3$exam10_3.potassium~as.factor(ex10_3$exam10_3.variety))
##
## Bartlett test of homogeneity of variances
##
## data: ex10_3$exam10_3.potassium by as.factor(ex10_3$exam10_3.variety)
## Bartlett's K-squared = 1.7512, df = 2, p-value = 0.4166
- Bartlett’s Test의 유의확률은 p-value = 0.4166으로 유의수준 0.05보다 매우 크므로 등분산성이 아니라는 대립가설을 채택할 근거가 없다.
- Runs Test와 Durbin-Watson test을 통하여 잔차의 자기상관성을 확인하여 보겠다.
Runs Test
anova_ex10_3 <- aov(ex10_3$exam10_3.potassium~as.factor(ex10_3$exam10_3.variety))
runs.test(anova_ex10_3$residuals, alternative = "two.sided", threshold = 0, plot = TRUE)
##
## Runs Test - Two sided
##
## data: anova_ex10_3$residuals
## Standardized Runs Statistic = -0.48591, p-value = 0.627
- Runs Test 결과 p-value = 0.6616이므로 귀무가설 “연속적인 관측값이 임의적이다.”를 기각하지 못한다.
Durbin-Watson test
dwtest(anova_ex10_3, alternative = "less")
##
## Durbin-Watson test
##
## data: anova_ex10_3
## DW = 1.7019, p-value = 0.8811
## alternative hypothesis: true autocorrelation is less than 0
- Durbin-Watson test의 경우 DW = 1.7019로써 0보다 2와 가깝게 나왔으며, 2보다 작으므로 alternative = “less” 주었고 p-value = 0.8811로 귀무가설 “자기상관이 0 이다.”를 기각하지 못한다.
- 자기상관성이 없다고 독립인 건 아니지만 독립성 가정이 만족된다고 가정할 수 있을 것 같다.
- Shapiro-Wilk test와 Bartlett’s Test으로 정규성과 등분산성을 확인하였으므로 ANOVA 검정을 진행한다.
summary(aov(ex10_3$exam10_3.potassium~as.factor(ex10_3$exam10_3.variety)))
## Df Sum Sq Mean Sq F value Pr(>F)
## as.factor(ex10_3$exam10_3.variety) 2 46.80 23.402 20.97 4.52e-05 ***
## Residuals 15 16.74 1.116
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
- p-value가 0.0000452으로 유의수준 0.05보다 매우 작기 때문에 귀무가설을 기각할 수 있고 따라서 유의수준 5%하에 그룹에 따라 밀의 칼륨 함유량의 모평균이 다르다고 말할 수 있다.
- 이 문제의 데이터는 정규성 가정과 등분산 가정을 만족하여 일원분산분석을 시행할 수 있지만, 문제에 나와 있는 대로 Welch’s test를 수행하도록 하겠다.
- Welch’s test란 표본의 정규성은 만족되지만 그룹별 분산이 동일하지 않은 경우에 매우 강력한 검정이다.
Welch’s test
library(onewaytests)
welch.test(ex10_3$exam10_3.potassium~as.factor(ex10_3$exam10_3.variety),data=ex10_3)
##
## Welch's Heteroscedastic F Test (alpha = 0.05)
## -------------------------------------------------------------
## data : ex10_3$exam10_3.potassium and as.factor(ex10_3$exam10_3.variety)
##
## statistic : 15.0096
## num df : 2
## denom df : 9.218254
## p.value : 0.00126072
##
## Result : Difference is statistically significant.
## -------------------------------------------------------------
- onewqy.test()에서 var.equal=F를 주면 같은 결과를 출력하여 준다.
oneway.test(ex10_3$exam10_3.potassium~as.factor(ex10_3$exam10_3.variety),var.equal=F)
##
## One-way analysis of means (not assuming equal variances)
##
## data: ex10_3$exam10_3.potassium and as.factor(ex10_3$exam10_3.variety)
## F = 15.01, num df = 2.0000, denom df = 9.2183, p-value = 0.001261
- Welch’s test 결과도 동일하게 p-value가 유의수준 0.05보다 매우 작기 때문에 귀무가설을 기각할 수 있고 따라서 유의수준 5%하에 그룹에 따라 밀의 칼륨 함유량의 모평균이 다르다고 말할 수 있다.
EXAMPLE 10.4


- 식물 뿌리 신장의 분산에 대한 제안된 분석은 4가지 화학 처리 각각에서 10개의 뿌리를 포함하는 것이다.
- 실험을 수행하고 실험에서 데이터를 수집하기 전에 제안된 시험의 검정력을 추정하는 것이 적절하고 바람직하다.
- 검정력을 계산할 때 대립가설 하에서 계산을 하게된다.
- 그 때 나오는 검정통계량은 \(\phi\) 를 따르게 되며 이를 구하는 공식및 결과는 다음과 같다.
- 문제에서 주어진 조건 하에서의 검정력을 구해 보자.
groups<-c(8,8,9,12)
power.anova.test(groups=4, n=10, between.var =var(groups), within.var = 7.5888, sig.level = 0.05, power=NULL)
##
## Balanced one-way analysis of variance power calculation
##
## groups = 4
## n = 10
## between.var = 3.583333
## within.var = 7.5888
## sig.level = 0.05
## power = 0.8621031
##
## NOTE: n is number in each group
- power=0.862로 높게 나왔다.
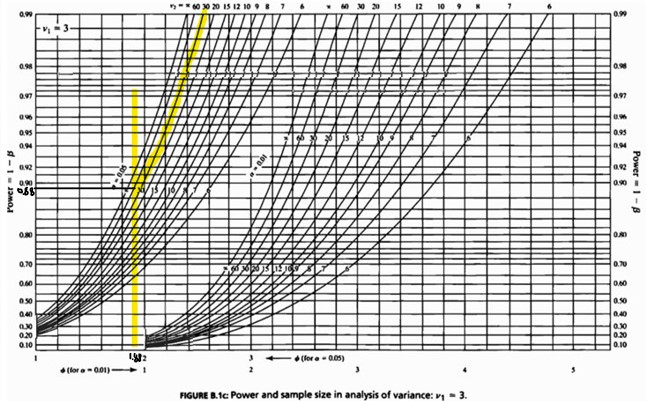
- 모집단 평균 간의 변동성이 지정된 경우 분산 분석의 검정력을 구할 수 있는 공식에 따라 \(\phi=1.88\) 나온다.
- 이를 이용해 power and sample size in analysis of variance: ν1=3 그래프에서 \(\phi=1.88\) 일때를 보면 그때의 검정력은 대략 0.88이고 이는 대립가설 하에서 제2종 오류를 범할 확률이 12%임을 의미한다.
EXAMPLE 10.5

- 최소 검출차가 주어졌을 때 분산의 검정력을 구하는 문제로써 구하는 공식은 다음과 같다.
공식을 함수로 작성하였다.
sample_size <- function(d,k,s,n){
phi=sqrt((n*d^2)/(2*k*s))
sample=data.frame(phi)
return(sample)
}
- 문제에서 주어진 조건하에 공식에 대입하여 보자.
- 작성한 sample_size 공식에 대입하여 \(\phi\)를 구하였다.
sample_size(d=4, k=4, s=7.5888, n=10)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.623411 |
- 다음은 pwr.1way 함수를 이용하여 power을 구하였다.
library(pwr2)
n <- 10
k <- 4
s2 <- 7.5888
delta<-4.0
phi<-round(sqrt((n*delta^2)/(2*k*s2)),2)
pwr.1way(k=4, n=10,f=NULL, alpha = 0.05, delta=4.0, sigma=sqrt(7.5888))
##
## Balanced one-way analysis of variance power calculation
##
## k = 4
## n = 10
## delta = 4
## sigma = 2.754778
## effect.size = 0.5133676
## sig.level = 0.05
## power = 0.7348545
##
## NOTE: n is number in each group, total sample = 40 power = 0.734854517715498
- power=0.735로 나왔다.
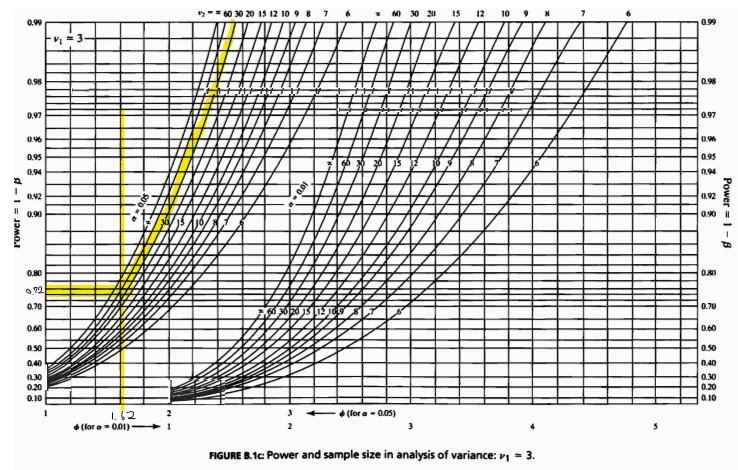
- 최소 검출 차가 지정된 경우 분산 분석의 검정력을 구할 수 있는 공식에 따라 \(\phi=1.62\)가 나온다
- 이를 이용해 power and sample size in analysis of variance: ν1=3 그래프에서 \(\phi=1.62\)일때를 보면 그때의 검정력은 대략 0.72이고 이는 대립가설 하에서 제2종 오류를 범할 확률이 28%임을 의미한다.
- 최소 탐지 가능한 차이가 지정된 경우 anova test의 power를 테스트하는 Balanced one-way analysis of variance power calculation 방법을 이용해 검정력을 구한 결과 또한 power=0.73으로 직접 구한 수치와 매우 유사한것을 볼 수 있다.
• 그룹 평균 간의 차이가 클수록 검정력은 커진다.
• 검정력은 표본 크기가 클수록 크다.
• 검정력은 더 적은 수의 그룹이 더 크다.
• 군내 변동성이 작을 경우 검정력이 더 크다.
• 검정력은 유의수준이 클수록 크다.
EXAMPLE 10.6


- One-Way Analysis of Variance 에서 필요한 샘플사이즈를 구해보도록 한다.
- 80%의 확률로 3.5kg 만큼의 차이를 탐지하기 위해 필요한 샘플사이즈를 계산하여 보겠다.
각 그룹에 대해 15만큼의 샘플이 필요하다고 가정하자
sample_size(d=3.5, k=4, s=9.383, n=15)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.56458 |
- 각 그룹에 대해 15만큼의 샘플이 필요하다고 가정하면 \(\phi=1.5645\)이고 표에서 찾아보면 이는 73%의 검정력에 해당한다.
각 그룹에 대해 20만큼의 샘플이 필요하다고 가정하자
sample_size(d=3.5, k=4, s=9.383, n=20)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.806622 |
- 각 그룹에 대해 20만큼의 샘플이 필요하다고 가정하면 \(\phi=1.80622\)이고 표에서 찾아보면 이는 84%의 검정력에 해당한다.
각 그룹에 대해 18만큼의 샘플이 필요하다고 가정하자
sample_size(d=3.5, k=4, s=9.383, n=18)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.713912 |
- 각 그룹에 대해 18만큼의 샘플이 필요하다고 가정하면 \(\phi=1.713912\)이고 표에서 찾아보면 이는 80%의 검정력에 거의 일치한다.
- 따라서 80% 이상의 검정력을 갖기 위해 필요한 최소한의 그룹별 샘플 사이즈는 18이라고 볼 수 있다.
EXAMPLE 10.7

- One-Way Analysis of Variance 에서 최소검출차를 구해보도록 한다.
- ex10_1의 데이터를 이용하여 최소 검출차를 추정하기 위해 다음과 같은 공식을 이용하자.
- 주어진 대로 공식을 함수로 작성하였다.
Minimum_Detectable_Difference <- function(k,s,p,n){
delta=sqrt((2*k*s*p^2)/n)
mdd = data.frame(delta)
return(mdd)
}
검정력: 90%, 그룹수: 4, 그룹별 샘플 수: 10, 유의수준: 0.05일 때 탐지할 수 있는 최소 검출차
Minimum_Detectable_Difference(k=4, s=9.3833, p=2.0, n=10)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| delta |
|---|
| 5.47965 |
- 검정력이 90%이고 그룹수는 4, 그룹별 샘플 수는 10, 유의수준은 0.05일 때 탐지할 수 있는 최소 검출차는 \(\delta=5.5\)로 계산되었다.
- 따라서 효과크기(최소검출차)=5.5일 때 집단간 차이가 유의하게 나타난다고 할 수 있다.
EXAMPLE 10.8

- ex10_1과 같은 실험을 생각하여 볼 때 다음과 같은 가정을 하여보자.
- 6개의 사료를 테스트할 수 있지만, 총 50마리의 돼지를 검사할 수 있는 공간과 장비만 가지고 있다고 하자.
유의수준 0.05, 80이상의 검정력으로 테스트하고 모집단 평균 간의 4.5kg의 작은 차이를 발견하고자 한다. - One-Way Analysis of Variance에서 검정력과 유의수준이 정해져 있을 때 필요한 최대 그룹수를 구하도록 해본다.
그룹 수 6으로 가정하자
sample_size(d=4.5, k=6, s=9.383, n=8)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.199488 |
- 그룹 수 6으로 가정하면 \(\phi=1.199488\)이다.
그룹 수 5로 가정하자
sample_size(d=4.5, k=5, s=9.383, n=10)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.469067 |
- 그룹 수 5로 가정하면 \(\phi=1.469067\)이다.
그룹 수 4로 가정하자
sample_size(d=4.5, k=4, s=9.383, n=12)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| phi |
|---|
| 1.799233 |
- 그룹 수 4로 가정하면 \(\phi=1.799233\)이다.
- 검정력이 80%일 때 \(\phi=1.8\)이므로 주어진 조건을 만족하기 위해서는 그룹 수를 4이하로 해야한다.
- k=4라고 가정하고 power를 산출하여 보면 0.80이므로 그룹 수는 4개라고 할 수 있다.
EXAMPLE 10.9


- 랜덤효과가 있는 분선분석의 검정력을 산출하는 공식은 다음과 같다.
- Example 10.2 데이터의 결과를 사용하여 ANOVA에서의 Random-Effect모델에서의 power를 구하여 보자.
random_effect<-function(v1, v2, s, alpha, ms){
F=((v2*s*qf(alpha, v1, v2, lower.tail = F))/((v2-2)*ms))
f=data.frame(F)
return(f)
}
- 계산 결과 F값은 1.54로 나왔으며, 이는 F분포표에서 자유도는 3과 16, 1-β 값이 0.1과 0.25 사이일 때 값임을 확인할 수 있다.
- 따라서 정확한 수치를 알수는 없지만 이들 사이의 값이 해당 분석의 검정력임을 알 수 있다.
random_effect(3, 16, 1.25, 0.05, 3.0)%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| F |
|---|
| 1.54232 |
F_value <- random_effect(3, 16, 1.25, 0.05, 3.0)[1,]
pf(F_value, 3, 16, lower.tail = F)
## [1] 0.2421396
- 이 랜덤효과 모델의 검정력은 약 0.24임을 알 수 있다.
EXAMPLE 10.10

#데이터셋
ex10_10%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_10.id | exam10_10.layer | exam10_10.abundance |
|---|---|---|
| 1 | 1 | 14.0 |
| 2 | 1 | 12.1 |
| 3 | 1 | 9.6 |
| 4 | 1 | 8.2 |
| 5 | 1 | 10.2 |
| 6 | 2 | 8.4 |
| 7 | 2 | 5.1 |
| 8 | 2 | 5.5 |
| 9 | 2 | 6.6 |
| 10 | 2 | 6.3 |
| 11 | 3 | 6.9 |
| 12 | 3 | 7.3 |
| 13 | 3 | 5.8 |
| 14 | 3 | 4.1 |
| 15 | 3 | 5.4 |
- 한 곤충학자는 낙엽 활엽수림에서 파리 종의 수직 분포를 연구하고 있으며,
세 가지 다른 식물 층인 허브, 관목, 나무에서 각각 다섯 개의 파리 모음을 얻는다. - 세 초목에 따른 파리들의 모이는 수의 분산에 차이가 있는지 검정해보도록 한다.
Shapiro-Wilk Test로 세 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_10$exam10_10.abundance,ex10_10$exam10_10.layer==1));shapiro.test(subset(ex10_10$exam10_10.abundance,ex10_10$exam10_10.layer==2));shapiro.test(subset(ex10_10$exam10_10.abundance,ex10_10$exam10_10.layer==3))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_10$exam10_10.abundance, ex10_10$exam10_10.layer == 1)
## W = 0.97015, p-value = 0.8762
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_10$exam10_10.abundance, ex10_10$exam10_10.layer == 2)
## W = 0.92329, p-value = 0.5514
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_10$exam10_10.abundance, ex10_10$exam10_10.layer == 3)
## W = 0.95891, p-value = 0.8004
- Shapiro-Wilk Test로 1,2,3 그룹 모두 정규성 검정한 결과 p-value가 0.05보다 매우 크므로 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 등분산성 검정으로 Bartlett’s Test를 진행하여 보자.
class(ex10_10$exam10_10.layer)
## [1] "numeric"
- layer의 class가 numeric이므로 factor로 변경해서 등분산성 검정을 실시한다.
Bartlett’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
bartlett.test(ex10_10$exam10_10.abundance~as.factor(ex10_10$exam10_10.layer))
##
## Bartlett test of homogeneity of variances
##
## data: ex10_10$exam10_10.abundance by as.factor(ex10_10$exam10_10.layer)
## Bartlett's K-squared = 1.7057, df = 2, p-value = 0.4262
- Bartlett’s Test의 유의확률은 p-value = 0.4262으로 유의수준 0.05보다 매우 크므로 등분산성이 아니라는 대립가설을 채택할 근거가 없다.
- Runs Test와 Durbin-Watson test을 통하여 잔차의 자기상관성을 확인하여 보겠다.
Runs Test
anova_ex10_10 <- aov(ex10_10$exam10_10.abundance~as.factor(ex10_10$exam10_10.layer))
runs.test(anova_ex10_10$residuals, alternative = "two.sided", threshold = 0, plot = TRUE)
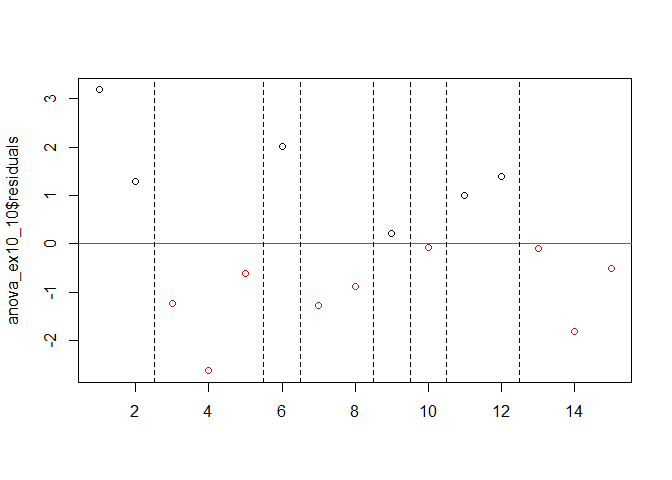
##
## Runs Test - Two sided
##
## data: anova_ex10_10$residuals
## Standardized Runs Statistic = -1.3282, p-value = 0.1841
- p-value = 0.2658로써 귀무가설을 기각하지 못하였다.
Durbin-Watson test
dwtest(anova_ex10_10, alternative = "less")
##
## Durbin-Watson test
##
## data: anova_ex10_10
## DW = 1.2926, p-value = 0.9829
## alternative hypothesis: true autocorrelation is less than 0
- Durbin-Watson test의 경우 DW = 1.2926로써 0보다 2와 가깝게 나왔으며, 2보다 작으므로 alternative = “less” 주었고 p-value = 0.9829로 귀무가설 “자기상관이 0 이다.”를 기각하지 못한다.
- 자기상관성이 없다고 독립인 건 아니지만 독립성 가정이 만족된다고 가정할 수 있을 것 같다.
- 정규성, 등분산성, 독립성이 만족되므로 ANOVA 검정을 실시한다.
summary(anova_ex10_10)
## Df Sum Sq Mean Sq F value Pr(>F)
## as.factor(ex10_10$exam10_10.layer) 2 73.58 36.79 13.18 0.000937 ***
## Residuals 12 33.50 2.79
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
- p-value는 0.0009로 유의수준보다 매우 작기 때문에 귀무가설을 기각할 수 있고 따라서 식물에 따라 파리의 개체수 분포가 다르다고 말할 수 있다.
- Kruskal-Wallis검정은 표본이 정규분포를 따르지 않지만 유사한 분포, 유사한 분산을 가질 때 적절한 비모수적 검정 방법이다.
kruskal.test(ex10_10$exam10_10.abundance~as.factor(ex10_10$exam10_10.layer))
##
## Kruskal-Wallis rank sum test
##
## data: ex10_10$exam10_10.abundance by as.factor(ex10_10$exam10_10.layer)
## Kruskal-Wallis chi-squared = 8.72, df = 2, p-value = 0.01278
- 같은 데이터에 대해 비모수적 방법, 즉 rank를 이용한 검정을 진행해 본 결과 p-value는 0.01278로써 유의수준보다 작기 때문에 귀무가설을 기각할 수 있다.
- 따라서 식물에 따라 파리의 개체수 분포가 다르다고 말할 수 있다.
library(pgirmess)
kruskalmc(ex10_10$exam10_10.abundance~as.factor(ex10_10$exam10_10.layer))
## Multiple comparison test after Kruskal-Wallis
## p.value: 0.05
## Comparisons
## obs.dif critical.dif difference
## 1-2 6.8 6.771197 TRUE
## 1-3 7.6 6.771197 TRUE
## 2-3 0.8 6.771197 FALSE
- Multiple comparison test after Kruskal-Wallis 실시한 결과 유의수준 5%하에 1-2, 1-3 그룹간 차이가 있다고 말할 수 있다.
EXAMPLE 10.11

#데이터셋
ex10_11%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_11.id | exam10_11.pond | exam10_11.ph |
|---|---|---|
| 1 | 1 | 7.68 |
| 2 | 1 | 7.69 |
| 3 | 1 | 7.70 |
| 4 | 1 | 7.70 |
| 5 | 1 | 7.72 |
| 6 | 1 | 7.73 |
| 7 | 1 | 7.73 |
| 8 | 1 | 7.76 |
| 9 | 2 | 7.71 |
| 10 | 2 | 7.73 |
| 11 | 2 | 7.74 |
| 12 | 2 | 7.74 |
| 13 | 2 | 7.78 |
| 14 | 2 | 7.78 |
| 15 | 2 | 7.80 |
| 16 | 2 | 7.81 |
| 17 | 3 | 7.74 |
| 18 | 3 | 7.75 |
| 19 | 3 | 7.77 |
| 20 | 3 | 7.78 |
| 21 | 3 | 7.80 |
| 22 | 3 | 7.81 |
| 23 | 3 | 7.84 |
| 24 | 4 | 7.71 |
| 25 | 4 | 7.71 |
| 26 | 4 | 7.74 |
| 27 | 4 | 7.79 |
| 28 | 4 | 7.81 |
| 29 | 4 | 7.85 |
| 30 | 4 | 7.87 |
| 31 | 4 | 7.91 |
- 이 예제는 데이터 정렬 후 tied rank 데이터가 있는 비모수 anova 검정을 사용하는 예제이다.
- 데이터는 4개의 연못에서의 산성도를 측정한 데이터이다.
- 가설은 다음과 같다.
- 네 연못의 pH농도가 같은지 검정하기 위해 먼저 그룹별 정규성 검정을 한다.
Shapiro-Wilk Test로 세 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_11$exam10_11.ph,ex10_11$exam10_11.pond==1));shapiro.test(subset(ex10_11$exam10_11.ph,ex10_11$exam10_11.pond==2));shapiro.test(subset(ex10_11$exam10_11.ph,ex10_11$exam10_11.pond==3));shapiro.test(subset(ex10_11$exam10_11.ph,ex10_11$exam10_11.pond==4))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_11$exam10_11.ph, ex10_11$exam10_11.pond == 1)
## W = 0.95, p-value = 0.7113
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_11$exam10_11.ph, ex10_11$exam10_11.pond == 2)
## W = 0.92959, p-value = 0.5123
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_11$exam10_11.ph, ex10_11$exam10_11.pond == 3)
## W = 0.97382, p-value = 0.9245
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_11$exam10_11.ph, ex10_11$exam10_11.pond == 4)
## W = 0.93368, p-value = 0.5502
- Shapiro-Wilk Test로 1,2,3,4 그룹 모두 정규성 검정한 결과 p-value가 0.05보다 매우 크므로 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 등분산성 검정으로 Bartlett’s Test를 진행하여 보자.
class(ex10_11$exam10_11.pond)
## [1] "numeric"
- pond의 class가 numeric이므로 factor로 변경해서 등분산성 검정을 실시한다.
Bartlett’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
bartlett.test(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond))
##
## Bartlett test of homogeneity of variances
##
## data: ex10_11$exam10_11.ph by as.factor(ex10_11$exam10_11.pond)
## Bartlett's K-squared = 8.8272, df = 3, p-value = 0.03168
- Bartlett’s Test의 유의확률은 p-value = 0.03168으로 유의수준 0.05보다 작으므로 등분산성이 아니라는 대립가설을 채택할 근거가 있다.
- 따라서 이 예제는 등분산성 가정을 만족하지 못한다.
- 등분산성 가정이 위배된 경우의 ANOVA test로 Welch’s test와 Brown and Forsythe 방법을 사용한다.
Welch’s test는 그룹별 표본크기는 동일하나 모분산이 동일하지 않은 경우 더 우수하지만 데이터의 분포가 매우 치우친 분포인 경우 귀무가설을 쉽게 기각하는 단점이 있다.
Brown and Forsythe 방법은 극단적인 평균값이 큰 분산과 관련 있는 경우에 검정력이 더 크다.
- 이 예제의 경우 Kruskal-Wallis test로 진행하였으므로 Kruskal-Wallis test를 진행하여 보자.
등분산성이 만족되지 않고 그룹별 샘플 수가 다르므로 비모수적 방법인 Kruskal-Wallis test 진행하여보자.
kruskal.test(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond))
##
## Kruskal-Wallis rank sum test
##
## data: ex10_11$exam10_11.ph by as.factor(ex10_11$exam10_11.pond)
## Kruskal-Wallis chi-squared = 11.944, df = 3, p-value = 0.007579
- p=0.0076로 귀무가설을 기각하므로 PH는 모든 네 개 연못이 같다고 할 수 없다.
- Kruskal-Wallis test에서 처리간의 차이가 있다고 할 때, 추가적으로 어느 처리에서 차이가 있는지 확인하여보자.
- R에서 pgirmess 패키지의 kruskalmc() 함수를 사용하여 순위합 개념을 이용한 다중비교 검정을 하겠다.
kruskalmc(ex10_11$exam10_11.ph, as.factor(ex10_11$exam10_11.pond))
## Multiple comparison test after Kruskal-Wallis
## p.value: 0.05
## Comparisons
## obs.dif critical.dif difference
## 1-2 9.6875000 11.99368 FALSE
## 1-3 13.8392857 12.41464 TRUE
## 1-4 13.5625000 11.99368 TRUE
## 2-3 4.1517857 12.41464 FALSE
## 2-4 3.8750000 11.99368 FALSE
## 3-4 0.2767857 12.41464 FALSE
- 결과를 확인하여 보면, 유의수준 0.05하에서 1-3, 1-4 차이가 있다고 할 수 있다.
- 4 그룹의 분산을 각각 확인하여 보면,
var(ex10_11[ex10_11$exam10_11.pond==1,3]);var(ex10_11[ex10_11$exam10_11.pond==2,3]);var(ex10_11[ex10_11$exam10_11.pond==3,3]);var(ex10_11[ex10_11$exam10_11.pond==4,3])
## [1] 0.0006839286
## [1] 0.001298214
## [1] 0.001228571
## [1] 0.005641071
- 각 그룹별 분산의 크기가 매우 작은 것을 볼 수 있다.
- 등분산성 가정이 위배되고 낮은 분산을 취했으므로 Welch’s test 비모수적 검정을 통하여 네 연못의 모평균 pH가 같은지 여부를 검정하여 보겠다.
welch.test(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond),data=ex10_11)
##
## Welch's Heteroscedastic F Test (alpha = 0.05)
## -------------------------------------------------------------
## data : ex10_11$exam10_11.ph and as.factor(ex10_11$exam10_11.pond)
##
## statistic : 7.898356
## num df : 3
## denom df : 14.37493
## p.value : 0.002369379
##
## Result : Difference is statistically significant.
## -------------------------------------------------------------
- p-value가 유의수준 0.05보다 작기 때문에 귀무가설을 기각할 수 있으며 연못에 따라 pH농도의 모평균 차이가 있다고 말할 수 있다.
- 등분산성이 만족되지 않고 그룹별 샘플 수가 다를 때 사용할 수 있는 사후검정법으로 Dunnett’s C test를 진행하여보겠다.
- R에서 DescTools 패키지의 DunnettTest() 함수를 사용하겠다.
library(DescTools)
DunnettTest(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond),control=1);DunnettTest(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond),control=2);DunnettTest(ex10_11$exam10_11.ph~as.factor(ex10_11$exam10_11.pond),control=3)
##
## Dunnett's test for comparing several treatments with a control :
## 95% family-wise confidence level
##
## $`1`
## diff lwr.ci upr.ci pval
## 2-1 0.04750000 -0.011567833 0.1065678 0.1360
## 3-1 0.07053571 0.009394699 0.1316767 0.0211 *
## 4-1 0.08500000 0.025932167 0.1440678 0.0037 **
##
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Dunnett's test for comparing several treatments with a control :
## 95% family-wise confidence level
##
## $`2`
## diff lwr.ci upr.ci pval
## 1-2 -0.04750000 -0.10656783 0.01156783 0.1359
## 3-2 0.02303571 -0.03810530 0.08417673 0.6748
## 4-2 0.03750000 -0.02156783 0.09656783 0.2866
##
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Dunnett's test for comparing several treatments with a control :
## 95% family-wise confidence level
##
## $`3`
## diff lwr.ci upr.ci pval
## 1-3 -0.07053571 -0.13142822 -0.009643206 0.0203 *
## 2-3 -0.02303571 -0.08392822 0.037856794 0.6644
## 4-3 0.01446429 -0.04642822 0.075356794 0.8802
##
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
- 결과를 확인하여 보면 그룹 3과 그룹1, 그룹4와 그룹1의 p-value가 0.05보다 작게 나왔다.
- 따라서 연못1과 연못3, 그리고 연못1과 연못4의 모평균 pH농도가 다르다고 말할 수 있다.
EXAMPLE 10.12
#데이터셋
ex10_12%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_12.id | exam10_12.side | exam10_12.height |
|---|---|---|
| 1 | 1 | 7.1 |
| 2 | 1 | 7.2 |
| 3 | 1 | 7.4 |
| 4 | 1 | 7.6 |
| 5 | 1 | 7.6 |
| 6 | 1 | 7.7 |
| 7 | 1 | 7.7 |
| 8 | 1 | 7.9 |
| 9 | 1 | 8.1 |
| 10 | 1 | 8.4 |
| 11 | 1 | 8.5 |
| 12 | 1 | 8.8 |
| 13 | 2 | 6.9 |
| 14 | 2 | 7.0 |
| 15 | 2 | 7.1 |
| 16 | 2 | 7.2 |
| 17 | 2 | 7.3 |
| 18 | 2 | 7.3 |
| 19 | 2 | 7.4 |
| 20 | 2 | 7.6 |
| 21 | 2 | 7.8 |
| 22 | 2 | 8.1 |
| 23 | 2 | 8.3 |
| 24 | 2 | 8.5 |
| 25 | 3 | 7.8 |
| 26 | 3 | 7.9 |
| 27 | 3 | 8.1 |
| 28 | 3 | 8.3 |
| 29 | 3 | 8.3 |
| 30 | 3 | 8.4 |
| 31 | 3 | 8.4 |
| 32 | 3 | 8.4 |
| 33 | 3 | 8.6 |
| 34 | 3 | 8.9 |
| 35 | 3 | 9.2 |
| 36 | 3 | 9.4 |
| 37 | 4 | 6.4 |
| 38 | 4 | 6.6 |
| 39 | 4 | 6.7 |
| 40 | 4 | 7.1 |
| 41 | 4 | 7.6 |
| 42 | 4 | 7.8 |
| 43 | 4 | 8.2 |
| 44 | 4 | 8.4 |
| 45 | 4 | 8.6 |
| 46 | 4 | 8.7 |
| 47 | 4 | 8.8 |
| 48 | 4 | 8.9 |
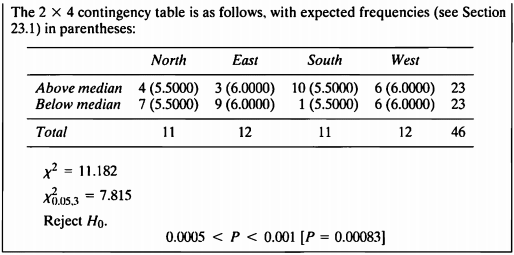
- 이 예제는 다표본 중위수 검정에 대한 예제이다.
- 데이터는 느릅나무의 길이를 건물의 4면에서 보았을 때의 높이를 기록한 데이터이다.
- 가설은 다음과 같다.
- 느릅나무 높이의 중앙값이 건물의 4면 모두에서 동일한지 검정하기 위해 먼저 각 샘플에 대해 정규성 검정을 진행한다.
Shapiro-Wilk Test로 세 그룹의 정규성을 평가하였다.
shapiro.test(subset(ex10_12$exam10_12.height,ex10_12$exam10_12.side==1));shapiro.test(subset(ex10_12$exam10_12.height,ex10_12$exam10_12.side==2));shapiro.test(subset(ex10_12$exam10_12.height,ex10_12$exam10_12.side==3));shapiro.test(subset(ex10_12$exam10_12.height,ex10_12$exam10_12.side==4))
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_12$exam10_12.height, ex10_12$exam10_12.side == 1)
## W = 0.95381, p-value = 0.6932
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_12$exam10_12.height, ex10_12$exam10_12.side == 2)
## W = 0.91943, p-value = 0.2812
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_12$exam10_12.height, ex10_12$exam10_12.side == 3)
## W = 0.93364, p-value = 0.4203
##
## Shapiro-Wilk normality test
##
## data: subset(ex10_12$exam10_12.height, ex10_12$exam10_12.side == 4)
## W = 0.89877, p-value = 0.1529
- Shapiro-Wilk Test로 1,2,3,4 그룹 모두 정규성 검정한 결과 p-value가 0.05보다 매우 크므로 모집단이 정규성을 따르지 않는다는 대립가설을 채택할 충분한 근거가 없다.
- 등분산성 검정으로 Bartlett’s Test를 진행하여 보자.
class(ex10_12$exam10_12.side)
## [1] "numeric"
- side의 class가 numeric이므로 factor로 변경해서 등분산성 검정을 실시한다.
Bartlett’s Test를 통해 네 그룹의 등분산성 검정을 진행하였다.
bartlett.test(ex10_12$exam10_12.height~as.factor(ex10_12$exam10_12.side))
##
## Bartlett test of homogeneity of variances
##
## data: ex10_12$exam10_12.height by as.factor(ex10_12$exam10_12.side)
## Bartlett's K-squared = 6.4423, df = 3, p-value = 0.09196
- Bartlett’s Test의 유의확률은 p-value = 0.09196으로 유의수준 0.05보다 크므로 등분산성이 아니라는 대립가설을 채택할 근거가 없다.
- 따라서 이 예제는 등분산성 가정을 만족한다.
- 중앙값에 대한 검정 중 비모수적인 방법인 Mood’s median test를 수행하도록 한다.
- R에서 RVAideMemoire패키지의 mood.medtest() 함수를 사용하였다.
median=median(ex10_12$exam10_12.height)
ex10_12$height <- ex10_12$exam10_12.height # height의 중앙값을 NA로 만들어줄 변수 생성
ex10_12$height[ex10_12$exam10_12.height == median]<-NA # height의 중앙값을 NA로 변경
ex10_12_na_rm<-na.omit(ex10_12) # height의 중앙값 7.9 제거
mood.medtest(ex10_12_na_rm$height~ex10_12_na_rm$exam10_12.side, data=ex10_12_na_rm, exact=F)
##
## Mood's median test
##
## data: ex10_12_na_rm$height by ex10_12_na_rm$exam10_12.side
## X-squared = 11.182, df = 3, p-value = 0.01078
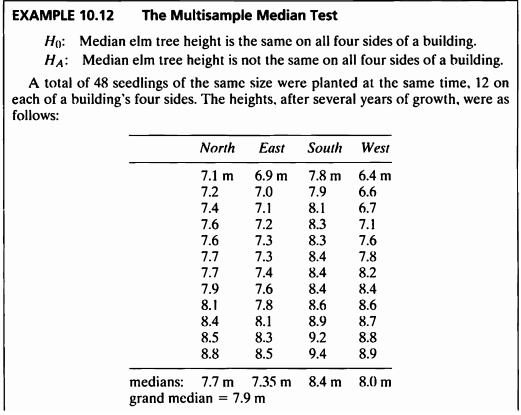
- Mood’s median test 검정 결과 p-value=0.01078으로 유의수준 0.05하에 귀무가설을 기각할 충분한 근거가 있다.
- 따라서 건물 4면에 서 보는 방향에 따라 느릅나무의 길이에 대한 중앙값의 분포에 차이가 있다는 충분한 근거가 있다.
카이제곱 검정
- 해당 문제는 분할표자료에서 카이제곱 검정을 수행해도 된다.
- 검정과정
모든 관측치를 통합한 수 중위수를 구한다.
중위수보다 크거나 작은 관측치의 개수를 센다.
분할표를 작성한다.
카이제곱 검정을 한다.
| North | East | South | West | ||
|---|---|---|---|---|---|
| Above median | 4(5.5) | 3(6.0) | 10(5.5) | 6(6.0) | 23 |
| Not above median | 7(5.5) | 9(6.0) | 1(5.5) | 6(6.0) | 23 |
| Total | 11 | 12 | 11 | 12 | 46 |
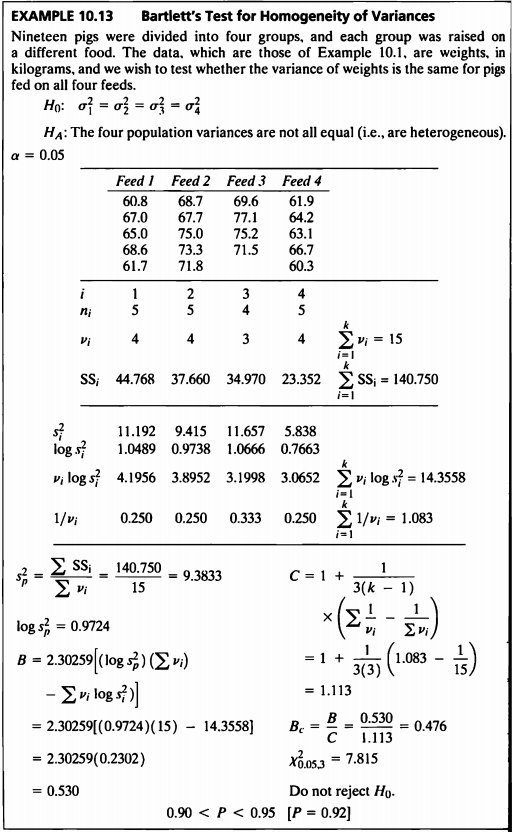
EXAMPLE 10.13
#데이터셋
ex10_13%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam10_13.id | exam10_13.diet | exam10_13.weight |
|---|---|---|
| 1 | 1 | 60.8 |
| 2 | 1 | 67.0 |
| 3 | 1 | 65.0 |
| 4 | 1 | 68.6 |
| 5 | 1 | 61.7 |
| 6 | 2 | 68.7 |
| 7 | 2 | 67.7 |
| 8 | 2 | 75.0 |
| 9 | 2 | 73.3 |
| 10 | 2 | 71.8 |
| 11 | 3 | 69.6 |
| 12 | 3 | 77.1 |
| 13 | 3 | 75.2 |
| 14 | 3 | 71.5 |
| 15 | 3 | NA |
| 16 | 4 | 61.9 |
| 17 | 4 | 64.2 |
| 18 | 4 | 63.1 |
| 19 | 4 | 66.7 |
| 20 | 4 | 60.3 |
- 이 예제는 Example 10.1 데이터를 사용하여 분산의 동질성 검정을 하는 예제이다.
- Example 10.1에서 보았듯이 네 그룹 모두 정규성 검정 결과 p-value가 0.05보다 매우 컸고 따라서 정규성을 만족하지 않는다고 볼만한 충분한 근거가 없었다.
- 정규성을 만족한다는 사실을 바탕으로 그룹별 등분산성 검정인 Bartlett’s Test를 진행한 결과 p-value가 0.9243으로 유의수준 0.05보다 매우 큰 수치이므로 귀무가설을 기각할 수 없고 따라서 네 그룹의 분산이 다르다고 볼만한 충분한 근거가 없다.
EXAMPLE 10-14


- 이 예제는 Example 10.1 데이터를 사용하여 변동계수에 대한 동질성 검정을 하는 예제이다.
- 본 검정의 가설은 다음과 같다.
- Homogeneity of Coefficients of variation 검정하는 함수를 작성하였다.
Homogeneity_CV<-function(n,m,ss){
v<-n-1
s<-sqrt(ss)
cv<-s/m
vv=sum(v*cv)
vp=sum(v*cv)/sum(v)
vp2=vp^2
vv2=sum(v*(cv)^2)
chisq=(vv2-(vv^2/sum(v)))/(vp2*(0.5+vp2))
pvalue=pchisq(chisq,3, lower.tail=F)
return(data.frame(chisq,pvalue))
}
Homogeneity_CV(tapply(ex10_1$exam10_1.weight,as.factor(ex10_1$exam10_1.diet), length), tapply(ex10_1$exam10_1.weight,as.factor(ex10_1$exam10_1.diet), mean), tapply(ex10_1$exam10_1.weight,as.factor(ex10_1$exam10_1.diet), var))%>%
kbl(caption = "Result") %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| chisq | pvalue |
|---|---|
| 0.3873096 | 0.9428514 |
- 그룹 수가 여러 개일 때 변동계수의 동일성 검정을 위해 주어진 공식을 사용하여 검정통계량을 구한 결과 0.37이었고, 임계값은 7.815였다.
- 따라서 귀무가설을 기각할 수 없고 그룹별 변동계수가 같지 않다고 말할만한 충분한 근거가 없다.
SAS 프로그램 결과
SAS 접기/펼치기 버튼
10장
LIBNAME ex 'C:\Biostat';
RUN;
/*10장 연습문제 불러오기*/
%MACRO chap10(name=,no=);
%do i=1 %to &no.;
PROC IMPORT DBMS=excel
DATAFILE="C:\Biostat\data_chap10"
OUT=ex.&name.&i. REPLACE;
RANGE="exam10_&i.$";
RUN;
%end;
%MEND;
%chap10(name=ex10_,no=13);
EXAMPLE 10.1
DATA exam_10_1;
SET ex.ex10_1;
if weight=. then delete;
RUN;
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_1 normal plot;
CLASS diet;
VAR weight;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_1 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*등분산성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_1;
CLASS diet;
MODEL weight=diet/p;
MEANS diet / HOVTEST=BARTLETT;
ods output Bartlett = Bartlett;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_1 : 등분산 가정 (Bartlett's Test for Homogeneity of weight Variance)";
PROC PRINT DATA=Bartlett label;
RUN;
title;
/*독립성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_1;
CLASS diet;
MODEL weight=diet/p;
output out=out_ds r=resid_var;
RUN;
DATA out_ds;
SET out_ds;
time=_n_;
ods graphics on;ods exclude none;ods results;
PROC GPLOT DATA=out_ds;
PLOT resid_var * time;
RUN;
/*anova*/
ods graphics off;ods exclude all;ods noresults;
PROC ANOVA data=ex.ex10_1;
class diet;
model weight=diet;
means diet/ TUKEY hovtest=levene(type=abs);
ods output OverallANOVA = OverallANOVA;
ods output CLDiffs=CLDiffs;
run;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=OverallANOVA;
RUN;
PROC PRINT DATA=CLDiffs;
RUN;
| OBS | VarName | diet | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | weight | 1 | Shapiro-Wilk | W | 0.933155 | Pr < W | 0.6180 | |
| 2 | weight | 2 | Shapiro-Wilk | W | 0.94389 | Pr < W | 0.6936 | |
| 3 | weight | 3 | Shapiro-Wilk | W | 0.952018 | Pr < W | 0.7287 | |
| 4 | weight | 4 | Shapiro-Wilk | W | 0.990268 | Pr < W | 0.9806 | |
| 5 | weight | 1 | Kolmogorov-Smirnov | D | 0.208622 | Pr > D | > | 0.1500 |
| 6 | weight | 2 | Kolmogorov-Smirnov | D | 0.2016 | Pr > D | > | 0.1500 |
| 7 | weight | 3 | Kolmogorov-Smirnov | D | 0.206041 | Pr > D | > | 0.1500 |
| 8 | weight | 4 | Kolmogorov-Smirnov | D | 0.145566 | Pr > D | > | 0.1500 |
| 9 | weight | 1 | Cramer-von Mises | W-Sq | 0.035311 | Pr > W-Sq | > | 0.2500 |
| 10 | weight | 2 | Cramer-von Mises | W-Sq | 0.033613 | Pr > W-Sq | > | 0.2500 |
| 11 | weight | 3 | Cramer-von Mises | W-Sq | 0.034212 | Pr > W-Sq | > | 0.2500 |
| 12 | weight | 4 | Cramer-von Mises | W-Sq | 0.020098 | Pr > W-Sq | > | 0.2500 |
| 13 | weight | 1 | Anderson-Darling | A-Sq | 0.235734 | Pr > A-Sq | > | 0.2500 |
| 14 | weight | 2 | Anderson-Darling | A-Sq | 0.220699 | Pr > A-Sq | > | 0.2500 |
| 15 | weight | 3 | Anderson-Darling | A-Sq | 0.215876 | Pr > A-Sq | > | 0.2500 |
| 16 | weight | 4 | Anderson-Darling | A-Sq | 0.151062 | Pr > A-Sq | > | 0.2500 |
| OBS | Effect | Dependent | Source | DF | Chi-Square | Pr > ChiSq |
|---|---|---|---|---|---|---|
| 1 | diet | weight | diet | 3 | 0.4752 | 0.9243 |
| OBS | Dependent | Source | DF | SS | MS | FValue | ProbF |
|---|---|---|---|---|---|---|---|
| 1 | weight | Model | 3 | 338.9373684 | 112.9791228 | 12.04 | 0.0003 |
| 2 | weight | Error | 15 | 140.7500000 | 9.3833333 | _ | _ |
| 3 | weight | Corrected Total | 18 | 479.6873684 | _ | _ | _ |
| OBS | Effect | Dependent | Method | Comparison | LowerCL | Difference | UpperCL | Significance |
|---|---|---|---|---|---|---|---|---|
| 1 | diet | weight | Tukey | 3 - 2 | -3.872 | 2.050 | 7.972 | 0 |
| 2 | diet | weight | Tukey | 3 - 1 | 2.808 | 8.730 | 14.652 | 1 |
| 3 | diet | weight | Tukey | 3 - 4 | 4.188 | 10.110 | 16.032 | 1 |
| 4 | diet | weight | Tukey | 2 - 3 | -7.972 | -2.050 | 3.872 | 0 |
| 5 | diet | weight | Tukey | 2 - 1 | 1.096 | 6.680 | 12.264 | 1 |
| 6 | diet | weight | Tukey | 2 - 4 | 2.476 | 8.060 | 13.644 | 1 |
| 7 | diet | weight | Tukey | 1 - 3 | -14.652 | -8.730 | -2.808 | 1 |
| 8 | diet | weight | Tukey | 1 - 2 | -12.264 | -6.680 | -1.096 | 1 |
| 9 | diet | weight | Tukey | 1 - 4 | -4.204 | 1.380 | 6.964 | 0 |
| 10 | diet | weight | Tukey | 4 - 3 | -16.032 | -10.110 | -4.188 | 1 |
| 11 | diet | weight | Tukey | 4 - 2 | -13.644 | -8.060 | -2.476 | 1 |
| 12 | diet | weight | Tukey | 4 - 1 | -6.964 | -1.380 | 4.204 | 0 |
PROC IML;
USE exam_10_1;
READ all;
CLOSE exam_10_1;
k= ncol(unique(diet));
N_total=nrow(id);
sum_group=repeat(0, k);
group_mean=repeat(0, k);
group_n=repeat(0, k);
%MACRO loc(group);
%do i=1 %to 4 %by 1;
feed&i. = loc( &group= &i);
sum_group[&i.] = sum(weight[feed&i.]);
group_mean[&i.] = mean(weight[feed&i.]);
group_n[&i.]=nrow(weight[feed&i.]);
%end;
%MEND;
%loc(diet);
*(a);
sum_total = sum(sum_group);
mean_total = round(sum_total/nrow(weight), 0.0001);
mean_vec = repeat(mean_total, N_total);
totalSS = round((weight-mean_vec)`*(weight-mean_vec), 0.0001);
totalDF= N_total-1;
groupSS_el = repeat(0,k);
%MACRO groupSS_el;
%do i=1 %to 4 %by 1;
groupSS_el[&i.] = round(group_n[&i.]*((group_mean[&i.]-mean_total)**2), 0.0001);
group_mean_vec&i. = repeat(group_mean[&i.], group_n[&i.])`;
%end;
%MEND;
%groupSS_el;
groupSS = sum(groupSS_el);
groupDF=k-1;
group_mean_vec=(group_mean_vec1 || group_mean_vec2 || group_mean_vec3 ||group_mean_vec4)`;
SSE = (weight-group_mean_vec)`*(weight-group_mean_vec);
within_DF =sum(group_n-repeat(1,k));
*(b);
group_SS_first_el=repeat(0,k);
%MACRO group_SS_first;
%do i=1 %to 4 %by 1;
group_SS_first_el[&i.] = (sum_group[&i.]**2)/group_n[&i.];
%end;
%MEND;
%group_SS_first;
group_SS_first = sum(group_SS_first_el);
sum_weight = sum(weight);
sum_weightSquare = weight`*weight;
totalDF = N_total-1;
groupsDF=k-1;
errorDF=N_total-k;
C=(sum_weight**2)/N_total;
b_totalSS = round(sum_weightSquare - C, 0.0001);
b_groupSS = round(group_SS_first - C, 0.0001);
b_errorSS = b_totalSS - b_groupSS;
*(c);
groupsMS = round( b_groupSS / groupsDF, 0.0001);
errorMS = round(b_errorSS/errorDF, 0.0001);
F = round(groupsMS /errorMS, 0.01);
critical_value = round(quantile('F', 1-0.05, groupsDF, errorDF), 0.01);
p_value = round(1- cdf('F', F, groupsDF, errorDF), 0.00001);
create exam10_1a_result var {totalSS totalDF groupSS groupDF SSE within_DF};
append;
close exam10_1a_result;
create exam10_1b_result var {b_totalSS b_groupSS b_errorSS};
append;
close exam10_1b_result;
create exam10_1c_result var {groupsMS errorMS F critical_value p_value};
append;
close exam10_1c_result;
title "Example 10.1";
PRINT group_n sum_group group_mean;
RUN;
QUIT;
| group_n | sum_group | group_mean |
|---|---|---|
| 5 | 323.1 | 64.62 |
| 5 | 356.5 | 71.3 |
| 4 | 293.4 | 73.35 |
| 5 | 316.2 | 63.24 |
EXAMPLE 10.1a
title "Example 10.1 (a)";
PROC PRINT DATA=exam10_1a_result;
RUN;
| OBS | TOTALSS | TOTALDF | GROUPSS | GROUPDF | SSE | WITHIN_DF |
|---|---|---|---|---|---|---|
| 1 | 479.687 | 18 | 338.937 | 3 | 140.75 | 15 |
EXAMPLE 10.1b
title "Example 10.1 (b)";
PROC PRINT DATA=exam10_1b_result;
RUN;
| OBS | B_TOTALSS | B_GROUPSS | B_ERRORSS |
|---|---|---|---|
| 1 | 479.687 | 338.937 | 140.75 |
EXAMPLE 10.1c
title "Example 10.1 (c)";
%MACRO loc(data, group, y_var);
%do i=1 %to 4 %by 1;
group&i. = loc( &group= &i);
sum_group[&i.] = sum(&y_var[group&i.]);
group_mean[&i.] = mean(&y_var[group&i.]);
group_n[&i.]=nrow(&y_var[group&i.]);
%end;
%MEND;
%MACRO group_SS_first;
%do i=1 %to 4 %by 1;
group_SS_first_el[&i.] = (sum_group[&i.]**2)/group_n[&i.];
%end;
%MEND;
%MACRO anova(data, group, y_var, SSdec, pvaluedec);
PROC IML;
USE &data;
READ all;
CLOSE &data;
k= ncol(unique(&group));
N=nrow(id);
sum_group=repeat(0, k);
group_mean=repeat(0, k);
group_n=repeat(0, k);
%loc(&data, &group, &y_var);
sum_&y_var. = sum(&y_var);
sumSquare_&y_var. = &y_var`*&y_var;
C = (sum_&y_var. **2)/N;
totalSS = round( sumSquare_&y_var. - C, 0.0001);
group_SS_first_el=repeat(0,k);
%group_SS_first;
group_SS_first = sum(group_SS_first_el);
groupsSS = group_SS_first - C;
errorSS = totalSS - groupsSS;
totalDF = N-1;
groupsDF = k-1;
errorDF = N-k;
groupsMS = round( groupsSS / groupsDF, &SSdec);
errorMS = round(errorSS/errorDF, &SSdec);
F = round(groupsMS /errorMS, 0.01);
critical_value = round(quantile('F', 1-0.05, groupsDF, errorDF), 0.01);
p_value = round(1- cdf('F', F, groupsDF, errorDF), &pvaluedec);
PRINT F critical_value p_value;
RUN;
QUIT;
%MEND;
%anova(exam_10_1, diet, weight, 0.0001, 0.00001);
| F | critical_value | p_value |
|---|---|---|
| 12.04 | 3.29 | 0.00028 |
EXAMPLE 10.2
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_2 normal plot;
CLASS technician;
VAR phosphorus;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_2 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*독립성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_2;
CLASS technician;
MODEL phosphorus=technician/p;
output out=out_ds r=resid_var;
RUN;
DATA out_ds;
SET out_ds;
time=_n_;
ods graphics on;ods exclude none;ods results;
PROC GPLOT DATA=out_ds;
PLOT resid_var * time;
RUN;
/*등분산성*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_2 plots=(DIAGNOSTICS RESIDUALS);
CLASS technician;
MODEL phosphorus=technician;
MEANS technician / HOVTEST=LEVENE(type=abs) hovtest=bf;
ods output HOVFTest = HOVFTest;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_2 : 등분산 가정 (Levene's Test & Brown and Forsythe's Test )";
PROC PRINT DATA=HOVFTest label;
RUN;
title;
/*KruskalWallisTest*/
ods graphics off;ods exclude all;ods noresults;
PROC NPAR1WAY DATA=ex.ex10_2 wilcoxon;
ods trace on;
CLASS technician;
VAR phosphorus;
ods output KruskalWallisTest=KruskalWallisTest;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_2 : KruskalWallisTest";
PROC PRINT DATA=KruskalWallisTest label;
RUN;
/*혼합모형 anova*/
ods graphics off;ods exclude all;ods noresults;
PROC MIXED DATA=ex.ex10_2 COVTEST CL;
CLASS technician ;
MODEL phosphorus= ;
RANDOM technician ;
ods output CovParms=CovParms;
RUN;
QUIT;
ods graphics on;ods exclude none;ods results;
title "ex10_2 : Covariance Parameter Estimates";
PROC PRINT DATA=CovParms label;
RUN;
title;
| OBS | VarName | technician | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | phosphorus | 1 | Shapiro-Wilk | W | 0.770908 | Pr < W | 0.0460 | |
| 2 | phosphorus | 2 | Shapiro-Wilk | W | 0.770908 | Pr < W | 0.0460 | |
| 3 | phosphorus | 3 | Shapiro-Wilk | W | 0.90202 | Pr < W | 0.4211 | |
| 4 | phosphorus | 4 | Shapiro-Wilk | W | 0.90202 | Pr < W | 0.4211 | |
| 5 | phosphorus | 1 | Kolmogorov-Smirnov | D | 0.348833 | Pr > D | 0.0441 | |
| 6 | phosphorus | 2 | Kolmogorov-Smirnov | D | 0.348833 | Pr > D | 0.0441 | |
| 7 | phosphorus | 3 | Kolmogorov-Smirnov | D | 0.221307 | Pr > D | > | 0.1500 |
| 8 | phosphorus | 4 | Kolmogorov-Smirnov | D | 0.221307 | Pr > D | > | 0.1500 |
| 9 | phosphorus | 1 | Cramer-von Mises | W-Sq | 0.10627 | Pr > W-Sq | 0.0707 | |
| 10 | phosphorus | 2 | Cramer-von Mises | W-Sq | 0.10627 | Pr > W-Sq | 0.0707 | |
| 11 | phosphorus | 3 | Cramer-von Mises | W-Sq | 0.042471 | Pr > W-Sq | > | 0.2500 |
| 12 | phosphorus | 4 | Cramer-von Mises | W-Sq | 0.042471 | Pr > W-Sq | > | 0.2500 |
| 13 | phosphorus | 1 | Anderson-Darling | A-Sq | 0.602789 | Pr > A-Sq | 0.0519 | |
| 14 | phosphorus | 2 | Anderson-Darling | A-Sq | 0.602789 | Pr > A-Sq | 0.0519 | |
| 15 | phosphorus | 3 | Anderson-Darling | A-Sq | 0.288595 | Pr > A-Sq | > | 0.2500 |
| 16 | phosphorus | 4 | Anderson-Darling | A-Sq | 0.288595 | Pr > A-Sq | > | 0.2500 |
| OBS | Effect | Dependent | Method | Source | DF | Sum of Squares | Mean Square | F Value | Pr > F |
|---|---|---|---|---|---|---|---|---|---|
| 1 | technician | phosphorus | LV | technician | 3 | 0.5120 | 0.1707 | 0.68 | 0.5755 |
| 2 | technician | phosphorus | LV | Error | 16 | 4.0000 | 0.2500 | _ | _ |
| 3 | technician | phosphorus | BF | technician | 3 | 0.8000 | 0.2667 | 0.41 | 0.7478 |
| 4 | technician | phosphorus | BF | Error | 16 | 10.4000 | 0.6500 | _ | _ |
| OBS | Variable | Chi-Square | Degrees of Freedom | Pr > Chi-Square |
|---|---|---|---|---|
| 1 | phosphorus | 6.0065 | 3 | 0.1113 |
| OBS | Cov Parm | Estimate | Standard Error | Z Value | Pr > Z | Alpha | Lower | Upper |
|---|---|---|---|---|---|---|---|---|
| 1 | technician | 0.3500 | 0.4978 | 0.70 | 0.2410 | 0.05 | 0.06931 | 384.71 |
| 2 | Residual | 1.2500 | 0.4419 | 2.83 | 0.0023 | 0.05 | 0.6934 | 2.8953 |
EXAMPLE 10.3
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_3 normal plot;
CLASS variety;
VAR potassium;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_3 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*welch's anova*/
ods graphics off;ods exclude all;ods noresults;
PROC ANOVA data=ex.ex10_3;
CLASS variety;
MODEL potassium=variety;
MEANS variety/ HOVTEST=BARTLETT welch;
ods output Bartlett = Bartlett;
ods output welch=welch;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_3 : 등분산 가정 (Bartlett's Test for Homogeneity of weight Variance)";
PROC PRINT DATA=Bartlett;
RUN;
title;
title "ex10_3 : Welch's test";
PROC PRINT DATA=welch;
RUN;
title;
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_3 ;
CLASS variety;
VAR potassium;
ods output moments=moments;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=moments;
RUN;
| OBS | VarName | variety | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | potassium | 1 | Shapiro-Wilk | W | 0.977796 | Pr < W | 0.9401 | |
| 2 | potassium | 2 | Shapiro-Wilk | W | 0.966512 | Pr < W | 0.8682 | |
| 3 | potassium | 3 | Shapiro-Wilk | W | 0.968814 | Pr < W | 0.8844 | |
| 4 | potassium | 1 | Kolmogorov-Smirnov | D | 0.176451 | Pr > D | > | 0.1500 |
| 5 | potassium | 2 | Kolmogorov-Smirnov | D | 0.184113 | Pr > D | > | 0.1500 |
| 6 | potassium | 3 | Kolmogorov-Smirnov | D | 0.151176 | Pr > D | > | 0.1500 |
| 7 | potassium | 1 | Cramer-von Mises | W-Sq | 0.028922 | Pr > W-Sq | > | 0.2500 |
| 8 | potassium | 2 | Cramer-von Mises | W-Sq | 0.032192 | Pr > W-Sq | > | 0.2500 |
| 9 | potassium | 3 | Cramer-von Mises | W-Sq | 0.023808 | Pr > W-Sq | > | 0.2500 |
| 10 | potassium | 1 | Anderson-Darling | A-Sq | 0.17631 | Pr > A-Sq | > | 0.2500 |
| 11 | potassium | 2 | Anderson-Darling | A-Sq | 0.205577 | Pr > A-Sq | > | 0.2500 |
| 12 | potassium | 3 | Anderson-Darling | A-Sq | 0.171315 | Pr > A-Sq | > | 0.2500 |
| OBS | Effect | Dependent | Source | DF | ChiSq | ProbChiSq |
|---|---|---|---|---|---|---|
| 1 | variety | potassium | variety | 2 | 1.7512 | 0.4166 |
| OBS | Effect | Dependent | Source | DF | FValue | ProbF |
|---|---|---|---|---|---|---|
| 1 | variety | potassium | variety | 2.0000 | 15.01 | 0.0013 |
| 2 | variety | potassium | Error | 9.2183 | _ | _ |
| OBS | VarName | variety | Label1 | cValue1 | nValue1 | Label2 | cValue2 | nValue2 |
|---|---|---|---|---|---|---|---|---|
| 1 | potassium | 1 | N | 6 | 6.000000 | 가중합 | 6 | 6.000000 |
| 2 | potassium | 1 | 평균 | 26.9833333 | 26.983333 | 관측값 합 | 161.9 | 161.900000 |
| 3 | potassium | 1 | 표준 편차 | 0.67946057 | 0.679461 | 분산 | 0.46166667 | 0.461667 |
| 4 | potassium | 1 | 왜도 | -0.1487695 | -0.148770 | 첨도 | -0.4470539 | -0.447054 |
| 5 | potassium | 1 | 제곱합 | 4370.91 | 4370.910000 | 수정 제곱합 | 2.30833333 | 2.308333 |
| 6 | potassium | 1 | 변동계수 | 2.518075 | 2.518075 | 평균의 표준 오차 | 0.27738862 | 0.277389 |
| 7 | potassium | 2 | N | 6 | 6.000000 | 가중합 | 6 | 6.000000 |
| 8 | potassium | 2 | 평균 | 25.6666667 | 25.666667 | 관측값 합 | 154 | 154.000000 |
| 9 | potassium | 2 | 표준 편차 | 1.13078144 | 1.130781 | 분산 | 1.27866667 | 1.278667 |
| 10 | potassium | 2 | 왜도 | 0.26299788 | 0.262998 | 첨도 | -0.0099899 | -0.009990 |
| 11 | potassium | 2 | 제곱합 | 3959.06 | 3959.060000 | 수정 제곱합 | 6.39333333 | 6.393333 |
| 12 | potassium | 2 | 변동계수 | 4.40564198 | 4.405642 | 평균의 표준 오차 | 0.46163959 | 0.461640 |
| 13 | potassium | 3 | N | 6 | 6.000000 | 가중합 | 6 | 6.000000 |
| 14 | potassium | 3 | 평균 | 29.55 | 29.550000 | 관측값 합 | 177.3 | 177.300000 |
| 15 | potassium | 3 | 표준 편차 | 1.26767504 | 1.267675 | 분산 | 1.607 | 1.607000 |
| 16 | potassium | 3 | 왜도 | -0.2292905 | -0.229290 | 첨도 | -0.9353331 | -0.935333 |
| 17 | potassium | 3 | 제곱합 | 5247.25 | 5247.250000 | 수정 제곱합 | 8.035 | 8.035000 |
| 18 | potassium | 3 | 변동계수 | 4.28993244 | 4.289932 | 평균의 표준 오차 | 0.51752617 | 0.517526 |
EXAMPLE 10.4
PROC POWER;
onewayanova
groupmeans=8.0 |8.0|9.0|12.0
stddev=2.754778
alpha=0.05
npergroup=10
power=. ;
RUN;
PROC IML;
k=4;
n=10;
v1 = k-1;
v2 = k*(n-1);
means_i = {8.0 8.0 9.0 12.0}`;
mu = mean(means_i);
sigma_2=7.5888;
phi = round(sqrt((n*((means_i - repeat(mu, 4))`*(means_i - repeat(mu, 4))))/(k*sigma_2)), 0.01);
PRINT phi v1 v2 ;
RUN;
QUIT;
The POWER Procedure
Overall F Test for One-Way ANOVA
| Fixed Scenario Elements | |
|---|---|
| Method | Exact |
| Alpha | 0.05 |
| Group Means | 8 8 9 12 |
| Standard Deviation | 2.754778 |
| Sample Size per Group | 10 |
| Computed Power |
|---|
| Power |
| 0.862 |
| phi | v1 | v2 |
|---|---|---|
| 1.88 | 3 | 36 |
EXAMPLE 10.5
PROC IML;
reset nolong;
k=4;d=4;s=7.5888;n=10;
p=sqrt((n*d*d)/(2*k*s));
print p;
RUN;
QUIT;
/*결과 p=1.5645802*/
| p |
|---|
| 1.6234108 |
EXAMPLE 10.6
PROC IML;
reset nolong;
k=4;d=3.5;s=9.383;n=15;
p=sqrt((n*d*d)/(2*k*s));
PRINT p;
RUN;
QUIT;
PROC IML;
reset nolong;
k=4;d=3.5;s=9.383;n=20;
p=sqrt((n*d*d)/(2*k*s));
PRINT p;
RUN;
QUIT;
PROC IML;
reset nolong;
k=4;d=3.5;s=9.383;n=18;
p=sqrt((n*d*d)/(2*k*s));
print p;
RUN;
QUIT;
/*결과 p=1.7139117*/
| p |
|---|
| 1.5645802 |
| p |
|---|
| 1.8066216 |
| p |
|---|
| 1.7139117 |
EXAMPLE 10.7
PROC IML;
reset nolong;
k=4;p=2;s=9.383;n=10;
d=sqrt((2*k*s*(p*p))/n);
print d;
RUN;
QUIT;
| d |
|---|
| 5.479562 |
EXAMPLE 10.8
PROC IML;
reset nolong;
d=4.5;s=9.383;
p=sqrt((8*d*d)/(2*6*s));/*k=6라 가정하고 시행*/
print p;
RUN;
QUIT;
PROC IML;
reset nolong;
d=4.5;s=9.383;
p=sqrt((10*d*d)/(2*5*s));/*k=5라 가정하고 시행*/
print p;
RUN;
QUIT;
PROC IML;
reset nolong;
d=4.5;s=9.383;
p=sqrt((12*d*d)/(2*4*s));/*k=4라 가정하고 시행*/
print p;
RUN;
QUIT;
/*k=4일때 power=0.80임을 알수있음*/
| p |
|---|
| 1.1994883 |
| p |
|---|
| 1.4690672 |
| p |
|---|
| 1.7992325 |
EXAMPLE 10.9
PROC IML;
reset nolong;
gr_ms=3;s=1.25;v1=3;v2=16;
F1=finv(1-0.05, v1,v2);
F2=(v2*s*F1)/((v2-2)*(gr_ms));
p=1-probF(F2,v1,v2);
print F1 F2 p;
RUN;
QUIT;
| F1 | F2 | p |
|---|---|---|
| 3.2388715 | 1.5423198 | 0.2421396 |
EXAMPLE 10.10
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_10 normal plot;
CLASS layer;
VAR abundance;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_10 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*등분산성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_10;
CLASS layer;
MODEL abundance=layer/p;
MEANS layer / HOVTEST=BARTLETT;
ods output Bartlett = Bartlett;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_10 : 등분산 가정 (Bartlett's Test for Homogeneity of weight Variance)";
PROC PRINT DATA=Bartlett label;
RUN;
title;
/*anova*/
ods graphics off;ods exclude all;ods noresults;
PROC ANOVA DATA=ex.ex10_10;
CLASS layer;
MODEL abundance = layer;
MEANS layer/ TUKEY hovtest=levene(type=abs);
ods output OverallANOVA = OverallANOVA;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=OverallANOVA;
RUN;
QUIT;
/*Kruskal-Wallis Test*/
ods graphics off;ods exclude all;ods noresults;
PROC NPAR1WAY DATA=ex.ex10_10;
CLASS layer;
VAR abundance;
ods output KruskalWallisTest=KruskalWallisTest;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_10 : Kruskal-Wallis Test";
PROC PRINT DATA=KruskalWallisTest;
RUN;
title;
QUIT;
| OBS | VarName | layer | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | abundance | 1 | Shapiro-Wilk | W | 0.970154 | Pr < W | 0.8762 | |
| 2 | abundance | 2 | Shapiro-Wilk | W | 0.923293 | Pr < W | 0.5514 | |
| 3 | abundance | 3 | Shapiro-Wilk | W | 0.958909 | Pr < W | 0.8004 | |
| 4 | abundance | 1 | Kolmogorov-Smirnov | D | 0.207939 | Pr > D | > | 0.1500 |
| 5 | abundance | 2 | Kolmogorov-Smirnov | D | 0.231739 | Pr > D | > | 0.1500 |
| 6 | abundance | 3 | Kolmogorov-Smirnov | D | 0.184327 | Pr > D | > | 0.1500 |
| 7 | abundance | 1 | Cramer-von Mises | W-Sq | 0.029496 | Pr > W-Sq | > | 0.2500 |
| 8 | abundance | 2 | Cramer-von Mises | W-Sq | 0.042843 | Pr > W-Sq | > | 0.2500 |
| 9 | abundance | 3 | Cramer-von Mises | W-Sq | 0.028686 | Pr > W-Sq | > | 0.2500 |
| 10 | abundance | 1 | Anderson-Darling | A-Sq | 0.189383 | Pr > A-Sq | > | 0.2500 |
| 11 | abundance | 2 | Anderson-Darling | A-Sq | 0.278219 | Pr > A-Sq | > | 0.2500 |
| 12 | abundance | 3 | Anderson-Darling | A-Sq | 0.200353 | Pr > A-Sq | > | 0.2500 |
| OBS | Effect | Dependent | Source | DF | Chi-Square | Pr > ChiSq |
|---|---|---|---|---|---|---|
| 1 | layer | abundance | layer | 2 | 1.7057 | 0.4262 |
| OBS | Dependent | Source | DF | SS | MS | FValue | ProbF |
|---|---|---|---|---|---|---|---|
| 1 | abundance | Model | 2 | 73.5840000 | 36.7920000 | 13.18 | 0.0009 |
| 2 | abundance | Error | 12 | 33.4960000 | 2.7913333 | _ | _ |
| 3 | abundance | Corrected Total | 14 | 107.0800000 | _ | _ | _ |
| OBS | Variable | ChiSquare | DF | Prob |
|---|---|---|---|---|
| 1 | abundance | 8.7200 | 2 | 0.0128 |
EXAMPLE 10.11
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_11 normal plot;
CLASS pond;
VAR ph;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_11 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*등분산성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_11;
CLASS pond;
MODEL ph=pond/p;
MEANS pond / HOVTEST=BARTLETT;
ods output Bartlett = Bartlett;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_11 : 등분산 가정 (Bartlett's Test for Homogeneity of weight Variance)";
PROC PRINT DATA=Bartlett label;
RUN;
title;
/*Kruskal-WallisTest*/
ods graphics off;ods exclude all;ods noresults;
PROC NPAR1WAY DATA=ex.ex10_11;
CLASS pond;
VAR ph;
ods output KruskalWallisTest=KruskalWallisTest;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_11 : Kruskal-Wallis Test";
PROC PRINT DATA=KruskalWallisTest;
RUN;
title;
QUIT;
/*welch's anova*/
ods graphics off;ods exclude all;ods noresults;
PROC ANOVA data=ex.ex10_11;
CLASS pond;
MODEL ph=pond ;
MEANS pond/ welch;
ods output welch=welch;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_11 : Welch's test";
PROC PRINT DATA=welch;
RUN;
title;
| OBS | VarName | pond | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | ph | 1 | Shapiro-Wilk | W | 0.950005 | Pr < W | 0.7113 | |
| 2 | ph | 2 | Shapiro-Wilk | W | 0.929589 | Pr < W | 0.5123 | |
| 3 | ph | 3 | Shapiro-Wilk | W | 0.97382 | Pr < W | 0.9245 | |
| 4 | ph | 4 | Shapiro-Wilk | W | 0.933682 | Pr < W | 0.5502 | |
| 5 | ph | 1 | Kolmogorov-Smirnov | D | 0.200477 | Pr > D | > | 0.1500 |
| 6 | ph | 2 | Kolmogorov-Smirnov | D | 0.222329 | Pr > D | > | 0.1500 |
| 7 | ph | 3 | Kolmogorov-Smirnov | D | 0.121718 | Pr > D | > | 0.1500 |
| 8 | ph | 4 | Kolmogorov-Smirnov | D | 0.157956 | Pr > D | > | 0.1500 |
| 9 | ph | 1 | Cramer-von Mises | W-Sq | 0.040945 | Pr > W-Sq | > | 0.2500 |
| 10 | ph | 2 | Cramer-von Mises | W-Sq | 0.058881 | Pr > W-Sq | > | 0.2500 |
| 11 | ph | 3 | Cramer-von Mises | W-Sq | 0.019496 | Pr > W-Sq | > | 0.2500 |
| 12 | ph | 4 | Cramer-von Mises | W-Sq | 0.032219 | Pr > W-Sq | > | 0.2500 |
| 13 | ph | 1 | Anderson-Darling | A-Sq | 0.255774 | Pr > A-Sq | > | 0.2500 |
| 14 | ph | 2 | Anderson-Darling | A-Sq | 0.325171 | Pr > A-Sq | > | 0.2500 |
| 15 | ph | 3 | Anderson-Darling | A-Sq | 0.150672 | Pr > A-Sq | > | 0.2500 |
| 16 | ph | 4 | Anderson-Darling | A-Sq | 0.24035 | Pr > A-Sq | > | 0.2500 |
| OBS | Effect | Dependent | Source | DF | Chi-Square | Pr > ChiSq |
|---|---|---|---|---|---|---|
| 1 | pond | ph | pond | 3 | 8.8272 | 0.0317 |
| OBS | Variable | ChiSquare | DF | Prob |
|---|---|---|---|---|
| 1 | ph | 11.9435 | 3 | 0.0076 |
| OBS | Effect | Dependent | Source | DF | FValue | ProbF |
|---|---|---|---|---|---|---|
| 1 | pond | ph | pond | 3.0000 | 7.90 | 0.0024 |
| 2 | pond | ph | Error | 14.3749 | _ | _ |
EXAMPLE 10.12
/*정규성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex10_12 normal plot;
CLASS side;
VAR height;
ods output TestsForNormality = TestsForNormality;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title " ex10_12 : 정규성 가정";
PROC PRINT DATA=TestsForNormality label;
RUN;
title;
/*등분산성 검정*/
ods graphics off;ods exclude all;ods noresults;
PROC GLM DATA=ex.ex10_12;
CLASS side;
MODEL height=side/p;
MEANS side/ HOVTEST=BARTLETT;
ods output Bartlett = Bartlett;
RUN;
ods graphics on;ods exclude none;ods results;
title "ex10_12 : 등분산 가정 (Bartlett's Test for Homogeneity of weight Variance)";
PROC PRINT DATA=Bartlett label;
RUN;
title;
PROC NPAR1WAY DATA=ex.ex10_12 median correct=no;
CLASS side;
VAR height;
RUN;
DATA e_1012;
INPUT median $ side $ ex_f;
CARDS;
above north 4
above east 3
above south 10
above west 6
below north 7
below east 9
below south 1
below west 6
;
RUN;
PROC FREQ DATA=e_1012 order=data;
WEIGHT ex_f;
TABLES median * side / nopercent nocol norow chisq;
RUN;
| OBS | VarName | side | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|---|
| 1 | height | 1 | Shapiro-Wilk | W | 0.953812 | Pr < W | 0.6932 | |
| 2 | height | 2 | Shapiro-Wilk | W | 0.919429 | Pr < W | 0.2812 | |
| 3 | height | 3 | Shapiro-Wilk | W | 0.933638 | Pr < W | 0.4203 | |
| 4 | height | 4 | Shapiro-Wilk | W | 0.898768 | Pr < W | 0.1529 | |
| 5 | height | 1 | Kolmogorov-Smirnov | D | 0.183335 | Pr > D | > | 0.1500 |
| 6 | height | 2 | Kolmogorov-Smirnov | D | 0.189739 | Pr > D | > | 0.1500 |
| 7 | height | 3 | Kolmogorov-Smirnov | D | 0.228172 | Pr > D | 0.0835 | |
| 8 | height | 4 | Kolmogorov-Smirnov | D | 0.161488 | Pr > D | > | 0.1500 |
| 9 | height | 1 | Cramer-von Mises | W-Sq | 0.046571 | Pr > W-Sq | > | 0.2500 |
| 10 | height | 2 | Cramer-von Mises | W-Sq | 0.070453 | Pr > W-Sq | > | 0.2500 |
| 11 | height | 3 | Cramer-von Mises | W-Sq | 0.074538 | Pr > W-Sq | 0.2286 | |
| 12 | height | 4 | Cramer-von Mises | W-Sq | 0.068918 | Pr > W-Sq | > | 0.2500 |
| 13 | height | 1 | Anderson-Darling | A-Sq | 0.26929 | Pr > A-Sq | > | 0.2500 |
| 14 | height | 2 | Anderson-Darling | A-Sq | 0.413744 | Pr > A-Sq | > | 0.2500 |
| 15 | height | 3 | Anderson-Darling | A-Sq | 0.399729 | Pr > A-Sq | > | 0.2500 |
| 16 | height | 4 | Anderson-Darling | A-Sq | 0.459638 | Pr > A-Sq | 0.2219 |
| OBS | Effect | Dependent | Source | DF | Chi-Square | Pr > ChiSq |
|---|---|---|---|---|---|---|
| 1 | side | height | side | 3 | 6.4423 | 0.0920 |
The NPAR1WAY Procedure
| Median Scores (Number of Points Above Median) for Variable height Classified by Variable side | |||||
|---|---|---|---|---|---|
| side | N | Sum of Scores | Expected Under H0 | Std Dev Under H0 | Mean Score |
| Average scores were used for ties. | |||||
| 1 | 12 | 4.50 | 6.0 | 1.483957 | 0.3750 |
| 2 | 12 | 3.00 | 6.0 | 1.483957 | 0.2500 |
| 3 | 12 | 10.50 | 6.0 | 1.483957 | 0.8750 |
| 4 | 12 | 6.00 | 6.0 | 1.483957 | 0.5000 |
| Median One-Way Analysis | ||
|---|---|---|
| Chi-Square | DF | Pr > ChiSq |
| 10.7283 | 3 | 0.0133 |
FREQ 프로시저
|
| ||||||||||||||||||||||||||||||||||||
median * side 테이블에 대한 통계량
| 통계량 | 자유도 | 값 | Prob |
|---|---|---|---|
| 카이제곱 | 3 | 11.1818 | 0.0108 |
| 우도비 카이제곱 | 3 | 12.5154 | 0.0058 |
| Mantel-Haenszel 카이제곱 | 1 | 2.4508 | 0.1175 |
| 파이 계수 | 0.4930 | ||
| 우발성 계수 | 0.4422 | ||
| 크래머의 V | 0.4930 |
표본 크기 = 46
EXAMPLE 10.13
ods graphics off;ods exclude all;ods noresults;
PROC ANOVA DATA=exam_10_1;
CLASS diet;
MODEL weight=diet;
MEANS diet/ hovtest= bartlett;
ods output Bartlett=Bartlett;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=Bartlett;
RUN;
| OBS | Effect | Dependent | Source | DF | ChiSq | ProbChiSq |
|---|---|---|---|---|---|---|
| 1 | diet | weight | diet | 3 | 0.4752 | 0.9243 |
EXAMPLE 10.14
PROC IML;
n={5, 5, 4, 5};
v={4, 4, 3, 4};
v_a={0.0518, 0.0597, 0.0465, 0.0456};
sum_v=sum(v);
sum_vv=sum(v#v_a);
v_p=sum_vv/sum_v;
v_p2=(v_p)**2;
sum_vv2=sum((v#v_a)#v_a);
chi_stat=round((sum_vv2-(((sum_vv)**2)/sum_v))/(v_p2*(0.5+v_p2)), 0.001);
chi_crit=round(abs(quantile("chisq", 1-0.05, 3)), 0.001);
PRINT sum_v sum_vv v_p v_p2 sum_vv2 chi_stat chi_crit;
RUN;
QUIT;
| sum_v | sum_vv | v_p | v_p2 | sum_vv2 | chi_stat | chi_crit |
|---|---|---|---|---|---|---|
| 15 | 0.7679 | 0.0511933 | 0.0026208 | 0.0397935 | 0.366 | 7.815 |
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
