[의학통계방법론] Ch9. Paired-Sample Hypotheses
Paired-Sample Hypotheses
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("readxl")
library("dplyr")
library("kableExtra")
library("PairedData")
엑셀파일불러오기
#모든 시트를 하나의 리스트로 불러오는 함수
read_excel_allsheets <- function(file, tibble = FALSE) {
sheets <- readxl::excel_sheets(file)
x <- lapply(sheets, function(X) readxl::read_excel(file, sheet = X))
if(!tibble) x <- lapply(x, as.data.frame)
names(x) <- sheets
x
}
9장
9장 연습문제 불러오기
#data_chap09에 연습문제 9장 모든 문제 저장
data_chap09 <- read_excel_allsheets("data_chap09.xls")
#연습문제 각각 데이터 생성
for (x in 1:length(data_chap09)){
assign(paste0('ex9_',1:length(data_chap09))[x],data_chap09[x])
}
#연습문제 데이터 형식을 리스트에서 데이터프레임으로 변환
for (x in 1:length(data_chap09)){
assign(paste0('ex9_',1:length(data_chap09))[x],data.frame(data_chap09[x]))
}
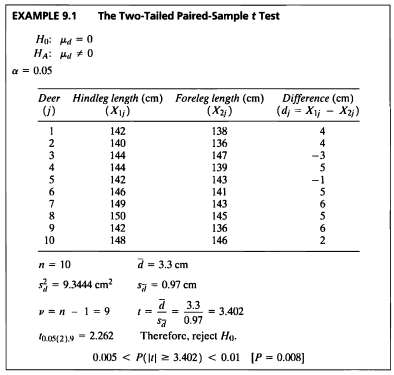
EXAMPLE 9.1
#데이터셋
ex9_1%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam9_1.deer | exam9_1.hindleg | exam9_1.foreleg |
|---|---|---|
| 1 | 142 | 138 |
| 2 | 140 | 136 |
| 3 | 144 | 147 |
| 4 | 144 | 139 |
| 5 | 142 | 143 |
| 6 | 146 | 141 |
| 7 | 149 | 143 |
| 8 | 150 | 145 |
| 9 | 142 | 136 |
| 10 | 148 | 146 |
- 해당 데이터는 사슴의 앞 다리와 뒷 다리에 대한 데이터로써 서로 짝을 이룬 대응표본 데이터이다.
- 사슴 모집단의 앞 다리 길이와 뒷 다리 길이의 차이가 있는지 검정하고자 한다.
- 서로 짝을 이룬 데이터이므로 Paired Sample t-test를 시행할 수 있으며, 앞 다리와 뒷 다리의 차이를 모수로 두어 One Sample t-test를 시행할 수도 있다.
- Paired Sample t-test의 경우 정규성을 만족해야하는 가정이 있으므로 정규성 검정을 시행한다.
표본의 크기가 작으므로 Shapiro-Wilk test를 사용할 것이다.
ex9_1$diff <- ex9_1$exam9_1.hindleg-ex9_1$exam9_1.foreleg
shapiro.test(ex9_1$diff)
##
## Shapiro-Wilk normality test
##
## data: ex9_1$diff
## W = 0.81366, p-value = 0.02123
- Paired Sample의 경우 정규성 가정시 두 표본에 대한 차이 값으로 정규성 검정을 시행하여야 한다.
- 정규성 검정의 결과 p-value = 0.02603으로써 정규성 가정을 만족하지 못하였으므로 비모수 검정을 시행하여야 하지만 비모수 검정은 EXAMPLE 9.4에서 다시 다루도록 하겠다.
- 이 예제에서는 Paired Sample t-test를 시행하여 보겠다.
t.test(ex9_1$exam9_1.hindleg,ex9_1$exam9_1.foreleg,mu=0,alt='two.sided', paired=T) # t.test(ex9_1$diff,mu=0,alt='two.sided') 같은 결과를 준다.
##
## Paired t-test
##
## data: ex9_1$exam9_1.hindleg and ex9_1$exam9_1.foreleg
## t = 3.4138, df = 9, p-value = 0.007703
## alternative hypothesis: true mean difference is not equal to 0
## 95 percent confidence interval:
## 1.113248 5.486752
## sample estimates:
## mean difference
## 3.3
- 검정통계량 값이 3.4138 문제의 값인 3.402와 다르게 나온 이유는 두 표본에 대한 표준편차가 반올림을 해서 계산이 되었기 때문이다.
- 이 예제에서는 두 표본에 대한 표준편차 0.9666667를 소수점 셋째자리에서 반올림하였다.
- p-value = 0.007703이므로 유의수준 5%하에 귀무가설을 기각할 수 있다.
- 즉, 사슴 모집단의 앞 다리와 뒷 다리의 길이의 차이가 있다고 할 수 있다.
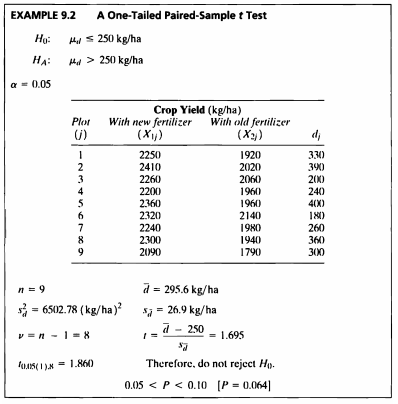
EXAMPLE 9.2
#데이터셋
ex9_2%>%
kbl(caption = "Dataset",escape=F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))
| exam9_2.plot | exam9_2.new | exam9_2.old | exam9_2.d |
|---|---|---|---|
| 1 | 2250 | 1920 | 330 |
| 2 | 2410 | 2020 | 390 |
| 3 | 2260 | 2060 | 200 |
| 4 | 2200 | 1960 | 240 |
| 5 | 2360 | 1960 | 400 |
| 6 | 2320 | 2140 | 180 |
| 7 | 2240 | 1980 | 260 |
| 8 | 2300 | 1940 | 360 |
| 9 | 2090 | 1790 | 300 |
- 해당 데이터는 기존 비료와 새로운 비료에 따른 수확량을 기록한 데이터이다.
- 새로운 비료와 기존 비료의 곡물 수확량 차이 모평균에 대한 검정을 수행할 것이다.
- Paired Sample t-test의 경우 정규성을 만족해야하는 가정이 있으므로 정규성 검정을 시행한다.
표본의 크기가 작으므로 Shapiro-Wilk test를 사용할 것이다.
shapiro.test(ex9_2$exam9_2.d)
##
## Shapiro-Wilk normality test
##
## data: ex9_2$exam9_2.d
## W = 0.94359, p-value = 0.6202
- p-value가 0.6202 이므로 유의수준 5%하에 두 집단 모두 정규성을 따르지 않는다고 할 충분한 증거가 없다.
- 따라서 정규성 가정을 만족한다 보고 Paired Sample t-test를 시행하겠다.
t.test(ex9_2$exam9_2.new, ex9_2$exam9_2.old, mu=250, alt='greater', paired=T) # t.test(ex9_2$exam9_2.d, mu=250, alt="greater") 같은 결과를 준다.
##
## Paired t-test
##
## data: ex9_2$exam9_2.new and ex9_2$exam9_2.old
## t = 1.6948, df = 8, p-value = 0.06428
## alternative hypothesis: true mean difference is greater than 250
## 95 percent confidence interval:
## 245.571 Inf
## sample estimates:
## mean difference
## 295.5556
EXAMPLE 9.3

- 이 예제는 Example 9.1 데이터를 사용하여 모분산이 같은지에 대한 검정을 해보도록 할 것이다.
- 두개의 paired-samples에 대해 분산이 같은지 여부를 검증하기 위해 Pitman 검정을 진행했다.
직접 함수를 작성해서 구하여 보자.
v1=round(var(ex9_1$exam9_1.hindleg),2)
v2=round(var(ex9_1$exam9_1.foreleg),2)
r=cor(ex9_1$exam9_1.hindleg,ex9_1$exam9_1.foreleg)
F=round(v1/v2,4)
n=length(ex9_1$exam9_1.hindleg)
t=round(((F-1)*sqrt(n-2))/(2*sqrt(F*(1-r^2))),3)
t_0.05=round(qt(0.025, df=8, lower.tail=F),3)
cat("\t F=",F," t=",t," t_0.05=",t_0.05, "\n\t Therefore cannot reject null hypothesis")
## F= 0.7111 t= -0.656 t_0.05= 2.306
## Therefore cannot reject null hypothesis
- t 값은 -0.656으로 자유도가 8인 t_0.05값 2.306보다 그 절대값이 작기 때문에 사슴 앞 다리의 길이와 사슴 뒷 다리의 길이의 모분산이 같다는 귀무가설을 기각할 수 없다.
다음은 PairedData 패키지의 pitman.morgan.test.default() 함수 사용
library(PairedData)
pitman.morgan.test.default(ex9_1$exam9_1.hindleg,ex9_1$exam9_1.foreleg)
##
## Paired Pitman-Morgan test
##
## data: ex9_1$exam9_1.hindleg and ex9_1$exam9_1.foreleg
## t = -0.65591, df = 8, p-value = 0.5303
## alternative hypothesis: true ratio of variances is not equal to 1
## 95 percent confidence interval:
## 0.227042 2.226964
## sample estimates:
## variance of x variance of y
## 11.56667 16.26667
- p-value가 0.5303으로 유의수준 5%하에 귀무가설을 기각할 수 없다.
Fisher의 방법으로 분산이 동일한지 검정
var.test(ex9_1$exam9_1.hindleg,ex9_1$exam9_1.foreleg,ratio=1,alternative = "two.sided",paired=T)
##
## F test to compare two variances
##
## data: ex9_1$exam9_1.hindleg and ex9_1$exam9_1.foreleg
## F = 0.71107, num df = 9, denom df = 9, p-value = 0.6197
## alternative hypothesis: true ratio of variances is not equal to 1
## 95 percent confidence interval:
## 0.1766186 2.8627458
## sample estimates:
## ratio of variances
## 0.7110656
- p-value가 0.6197로 유의수준 5%하에 귀무가설을 기각할 수 없다.
- 따라서 유의수준 5%하에 사슴 앞 다리 길이와 뒷 다리 길이의 모분산이 다르다고 말할만한 충분한 근거가 없다.
EXAMPLE 9.4
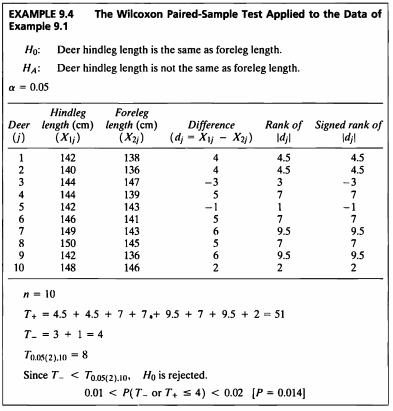
- 이 예제에서는 Example 9.1 데이터가 정규성 가정을 만족하지 못하였으므로 비모수 검정을 시행하여보겠다.
ex9_1$diff=ex9_1$exam9_1.hindleg-ex9_1$exam9_1.foreleg
ex9_1$d.rank= rank(abs(ex9_1$diff))
ex9_1$sign_rank= ifelse(ex9_1$diff < 0, ex9_1$d.rank *(-1), ex9_1$d.rank)
T.plus= sum(subset(ex9_1, sign_rank>=0)$sign_rank)
T.minus= abs(sum(subset(ex9_1, sign_rank< 0)$sign_rank))
T.plus;T.minus
## [1] 51
## [1] 4
- signed rank의 양수 합은 51, 음수 합은 4이다.
- 대응 표본에 대하여 비모수 검정법인 Wilcoxon signed rank test 수행하였다.
wilcox.test(ex9_1$exam9_1.hindleg, ex9_1$exam9_1.foreleg, paired=T)
##
## Wilcoxon signed rank test with continuity correction
##
## data: ex9_1$exam9_1.hindleg and ex9_1$exam9_1.foreleg
## V = 51, p-value = 0.01859
## alternative hypothesis: true location shift is not equal to 0
- p유의수준 5%에서 기각역은 8이다.
- 4 < 8이므로 귀무가설을 기각하고 대립가설을 채택한다.
- 즉. 유의수준 5% 하에서 사슴 앞다리 길이의 모평균과 뒷다리 길이의 모평균은 다르다고 볼 수 있는 충분한 근거가 있다.
SAS 프로그램 결과
SAS 접기/펼치기 버튼
9장
LIBNAME ex 'C:\Biostat';
RUN;
/*9장 연습문제 불러오기*/
%macro chap09(name=,no=);
%do i=1 %to &no.;
PROC IMPORT DBMS=excel
DATAFILE="C:\Biostat\data_chap09"
OUT=ex.&name.&i. REPLACE;
RANGE="exam9_&i.$";
RUN;
%end;
%mend;
%chap09(name=ex9_,no=2);
EXAMPLE 9.1
DATA ex9_1_diff;
SET ex.ex9_1;
diff=hindleg-foreleg;
abs_diff = abs(diff);
RUN;
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex9_1_diff normal;
VAR diff;
ods output TestsForNormality = TestsForNormality;
RUN;
PROC TTEST DATA=ex.ex9_1;
PAIRED hindleg * foreleg ;
ods output TTests =TTests;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title 'Example 9.1: 정규성검정';
PROC PRINT DATA=TestsForNormality label;
RUN;
title 'Example 9.1: Two-Tailed Paired-Sample t Test';
PROC PRINT DATA=TTests label;
RUN;
| OBS | VarName | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|
| 1 | diff | Shapiro-Wilk | W | 0.813656 | Pr < W | 0.0212 | |
| 2 | diff | Kolmogorov-Smirnov | D | 0.290563 | Pr > D | 0.0177 | |
| 3 | diff | Cramer-von Mises | W-Sq | 0.150917 | Pr > W-Sq | 0.0198 | |
| 4 | diff | Anderson-Darling | A-Sq | 0.831819 | Pr > A-Sq | 0.0213 |
| OBS | Variable 1 | Variable 2 | Difference | t Value | DF | Pr > |t| |
|---|---|---|---|---|---|---|
| 1 | hindleg | foreleg | hindleg - foreleg | 3.41 | 9 | 0.0077 |
EXAMPLE 9.2
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex.ex9_2 normal;
VAR d;
ods output TestsForNormality = TestsForNormality;
RU;
PROC TTEST order=data h0=250 SIDES=u
DATA=ex.ex9_2;
PAIRED new * old ;
ods output TTests =TTests;
RUN;
ods graphics on;ods exclude none;ods results;
PROC SORT DATA=TestsForNormality;
BY descending Test;
RUN;
title 'Example 9.2: 정규성검정';
PROC PRINT DATA=TestsForNormality label;
run;
title 'Example 9.2: One-Tailed Paired-Sample t Test';
PROC PRINT DATA=TTests label;
RUN;
| OBS | VarName | 적합도 검정 | 적합도 통계량에 대한 레이블 | 적합도 통계량 값 | p-값 레이블 | p-값의 부호 | p-값 |
|---|---|---|---|---|---|---|---|
| 1 | d | Shapiro-Wilk | W | 0.94359 | Pr < W | 0.6202 | |
| 2 | d | Kolmogorov-Smirnov | D | 0.121236 | Pr > D | > | 0.1500 |
| 3 | d | Cramer-von Mises | W-Sq | 0.028216 | Pr > W-Sq | > | 0.2500 |
| 4 | d | Anderson-Darling | A-Sq | 0.21151 | Pr > A-Sq | > | 0.2500 |
| OBS | Variable 1 | Variable 2 | Difference | t Value | DF | Pr > t |
|---|---|---|---|---|---|---|
| 1 | new | old | new - old | 1.69 | 8 | 0.0643 |
EXAMPLE 9.3
PROC IML;
use ex.ex9_1 ; read all;
n=10;
s1=var(hindleg);
s2=var(foreleg);
F=s1/s2;
r=0.6739;
t=((F-1)*sqrt(n-2))/(2*sqrt(F*(1-(r*r))));
p=probt(-0.655, 8)*2;
PRINT F t p;
RUN;
QUIT;
| F | t | p |
|---|---|---|
| 0.7110656 | -0.655872 | 0.530846 |
EXAMPLE 9.4
ods graphics off;ods exclude all;ods noresults;
PROC UNIVARIATE DATA=ex9_1_diff;
VAR diff;
ods output TestsForLocation = TestsForLocation;
RUN;
ods graphics on;ods exclude none;ods results;
PROC PRINT DATA=TestsForLocation label;
RUN;
| OBS | VarName | 위치모수 검정 | 검정 통계량 레이블 | 검정 통계량 | P-값 설명 | P-값 | Mu0 값 |
|---|---|---|---|---|---|---|---|
| 1 | diff | 스튜던트의 t | t | 3.413793 | Pr > |t| | 0.0077 | 0 |
| 2 | diff | 부호 | M | 3 | Pr >= |M| | 0.1094 | 0 |
| 3 | diff | 부호 순위 | S | 23.5 | Pr >= |S| | 0.0117 | 0 |
PROC RANK DATA=ex9_1_diff out=ex9_1_rank;
VAR abs_diff;
RANKS rank;
RUN;
DATA ex9_1_signed_rank;
SET ex9_1_rank;
if diff < 0 then signed_rank = rank*(-1);
else if diff > 0 then signed_rank = rank;
RUN;
PROC PRINT DATA=ex9_1_signed_rank;
RUN;
PROC IML;
use ex9_1_signed_rank;
read all;
close ex9_1_signed_rank;
n = nrow(deer);
plus = loc(diff>0);
minus = loc(diff<0);
rank_plus = rank[plus];
rank_minus = rank[minus];
T_plus = sum(rank_plus);
T_minus = sum(rank_minus);
PRINT T_plus T_minus ;
RUN;
QUIT;
| OBS | deer | hindleg | foreleg | diff | abs_diff | rank | signed_rank |
|---|---|---|---|---|---|---|---|
| 1 | 1 | 142 | 138 | 4 | 4 | 4.5 | 4.5 |
| 2 | 2 | 140 | 136 | 4 | 4 | 4.5 | 4.5 |
| 3 | 3 | 144 | 147 | -3 | 3 | 3.0 | -3.0 |
| 4 | 4 | 144 | 139 | 5 | 5 | 7.0 | 7.0 |
| 5 | 5 | 142 | 143 | -1 | 1 | 1.0 | -1.0 |
| 6 | 6 | 146 | 141 | 5 | 5 | 7.0 | 7.0 |
| 7 | 7 | 149 | 143 | 6 | 6 | 9.5 | 9.5 |
| 8 | 8 | 150 | 145 | 5 | 5 | 7.0 | 7.0 |
| 9 | 9 | 142 | 136 | 6 | 6 | 9.5 | 9.5 |
| 10 | 10 | 148 | 146 | 2 | 2 | 2.0 | 2.0 |
| T_plus | T_minus |
|---|---|
| 51 | 4 |
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
