[의학통계방법론] Ch3. Measures of Central Tendency
Measures of Central Tendency
정리내용 접기/펼치기 버튼
3.1 The Arithmetic Mean
- 산술평균 (arithmetic mean)
- 모든 관측값들의 합을 표본크기로 나눈 것
- 데이터는 구간 척도(interval scale), 비 척도(ratio scale)이어야 한다.
- 모든 값들이 계산에 이용된다.
- 유일하다.(unique)
- 평균으로부터의 편차의 합은 항상 0 이다.
- 이상치에 영향을 받는다.
- 가중평균 (weighted arithmetic mean)
- 서로 다른 표본크기를 가지는 표본들의 평균값을 구할 때 사용
3.2 The Median
- 중위수 / 중앙값 (median)
- 정렬된 자료에서 가장 가운데 위치한 수
- 이상치에 영향을 받지 않는다.
- 유일하다.(unique)
- 예) 1,2,6,7,8 (n=5, 홀수): 3번째 값 = 6.
- 예) 1,2,7,8,9,10 (n=6, 짝수): 3번째와 4번째 값의 평균 = 7.5
- 실제 예) 소득, 병원 입원기간 (데이터가 중앙에 몰려있는 자료)
- 도수분포표에서의 중위수 계산
- 누적 도수를 이용하여 중위수가 있을 구간을 찾는다.
- 구간 크기 (interval size)를 계산한다.
- 총 빈도(n)을 계산한다.
- 예상 구간 바로 전 구간까지의 누적빈도를 계산한다.
- 중심경향 (central tendency)의 측도는
- 이상치가 없다면 일반적으로 평균 사용
- 이상치가 있는 경우에는 이에 덜 민감한 중위수 사용
3.3 The Mode
- 최빈값 (mode)
- 가장 빈번하게 관측된 측정값
- 이상치에 영향을 받지 않는다.
- 수치형 자료나 범주형 자료 모두에 사용될 수 있다.
- 최빈값이 없을 수도 있다.
- 여러개의 최빈값이 존재할 수도 있다. (not unique)
3.4 Other Measures of Central Tendency
- 기하평균 (geometric mean; GM)
- 데이터가 양수이면서 오른쪽으로 치우친 경우 사용 (0 포함된 데이터 경우 사용 X)
- 데이터가 변화의 비(ratio of change)로 측정된 경우
- 두 그룹에 대하여 데이터의 범위(range)가 다른 경우
- 예: 인구 증가율
- 데이터가 같지 않다면 기하평균은 산술평균보다 항상 작다.
- 기하평균의 필요성
- 곱셈으로 계산하는 값에서의 평균을 계산하고자 할 때 기하 평균을 사용한다.
- 예를 들어 어떤 값이 처음에 1000이고, 첫 해에 10% 증가하고,그 다음 해 에 20% 증가하고, 그 다음 해에 15% 감소했다고 할 때 결과 값은 처음의 값 1000에 1.1,1.2, 0.85의 기하평균을 세 번 곱한 값이 된다. 1.1,1.2, 0.85 의 기하평균 (1.1 x 1.2 x 0.85)/3 = 1.0391… 이므로,3년동안 평균 3.91%씩 증가한 셈이다. 즉,1000 x 1.1 x 1.2 x 0.85 = 1000(1.0391)3이다.
- 조화평균 (harmonic mean; HM)
- 데이터가 양수이며 비척도(ratio scale)인 경우 사용
- 평균적인 변화율을 구할 때 주로 사용
- 데이터가 양수이고 같지 않다면 조화평균<기하평균<산술평균 이다.
- 조화평균의 필요성
- 평균적인 변화율을 구하고자 할 때 사용
- 예를 들어, 전체 거리의 절반을 40km/h의 속도로 달리고, 남은 절반을 60km/h 로 달렸다면,평균 속력은 40과 60의 조화 평균인 48km/h가 된다.
- 절단평균 (truncated mean, trimmed mean)
- 데이터의 양쪽 꼬리부분 즉, 작은 값이나 큰 값의 일부를 제외하고 구한 평균
- 보통 5% ~ 25%의 자료를 버리고 구한다.
- 특이치가 있는 경우 덜 민감한 평균을 계산할 수 있다.
- 분포가 대칭적이지 않을 경우에는 편향된 평균값을 계산할 수 있게 된다.
- 사분위평균 (interquartile mean; IQM)
- 데이터의 상위 25%, 하위 25%를 버리고 구한 평균
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("readxl")
library("ggplot2")
library("Hmisc")
library("psych")
엑셀파일불러오기
library('readxl')
#모든 시트를 하나의 리스트로 불러오는 함수
read_excel_allsheets <- function(file, tibble = FALSE) {
sheets <- readxl::excel_sheets(file)
x <- lapply(sheets, function(X) readxl::read_excel(file, sheet = X))
if(!tibble) x <- lapply(x, as.data.frame)
names(x) <- sheets
x
}
3장
3장 연습문제 불러오기
#data_chap03에 연습문제 3장 모든 문제 저장
data_chap03 <- read_excel_allsheets("data_chap03.xls")
#연습문제 각각 데이터 생성
for (x in 1:length(data_chap03)){
assign(paste0('ex3_',1:length(data_chap03))[x],data_chap03[x])
}
#연습문제 데이터 형식을 리스트에서 데이터프레임으로 변환
for (x in 1:length(data_chap03)){
assign(paste0('ex3_',1:length(data_chap03))[x],data.frame(data_chap03[x]))
}
EXAMPLE 3.1

#데이터셋
ex3_1
## Length
## 1 3.3
## 2 3.5
## 3 3.6
## 4 3.6
## 5 3.7
## 6 3.8
## 7 3.8
## 8 3.8
## 9 3.9
## 10 3.9
## 11 3.9
## 12 4.0
## 13 4.0
## 14 4.0
## 15 4.0
## 16 4.1
## 17 4.1
## 18 4.1
## 19 4.2
## 20 4.2
## 21 4.3
## 22 4.3
## 23 4.4
## 24 4.5
sum_x <- sum(ex3_1$Length) ; cat('Sum of X =', sum_x)
## Sum of X = 95
n <- length(ex3_1$Length); cat('Sample Size =', n)
## Sample Size = 24
x_bar <- mean(ex3_1$Length); cat('Mean of X =', x_bar)
## Mean of X = 3.958333
날개의 합은 95 cm 이며 평균은 3.96 cm 이다.
EXAMPLE 3.2

#데이터셋
ex3_2
## exam3_2.X exam3_2.Freq
## 1 3.3 1
## 2 3.4 0
## 3 3.5 1
## 4 3.6 2
## 5 3.7 1
## 6 3.8 3
## 7 3.9 3
## 8 4.0 4
## 9 4.1 3
## 10 4.2 2
## 11 4.3 2
## 12 4.4 1
## 13 4.5 1
- 가중평균 계산
library(Hmisc) # wtd.mean 사용하기 위해
xi <- ex3_2$exam3_2.X
fi <- ex3_2$exam3_2.Freq
wm <- wtd.mean(xi, weights=fi) #가중평균
round(wm,2)
## [1] 3.96
sum_fixi <- sum(fi*xi) # 가중합
sum_fi <- sum(fi) #가중치 합
w_mean<-sum_fixi/sum_fi #가중평균
cat("가중합 = ",sum_fixi," ","가중치 합 = ",sum_fi," ","가중평균 = ",w_mean)
## 가중합 = 95 가중치 합 = 24 가중평균 = 3.958333
- 중위수 계산
중위수는 정렬된 자료에서 중앙에 위치한 수이다.
도수분포표에서는 R에 내장되어 있는 함수인 wtd.quantile() 함수를 사용해서 중위수를 구할 수 있다.
x.median <- wtd.quantile(xi, weights=fi, probs=c(0,.25,.5,.75,1), type=c("i/n"))
wmed <- round(x.median,5)
wvar <- wtd.var(xi, weights=fi) # 분산
cat("wtd.quatile() 로 구한 중위수","\n","중위수 = ",wmed[3]," 분산 = ",wvar)
## wtd.quatile() 로 구한 중위수
## 중위수 = 3.925 분산 = 0.08514493
| \(X_i(cm)\) | \(f_i\) | \(f_iX_i(cm)\) |
|---|---|---|
| 3.3 | 1 | 3.3 |
| 3.4 | 0 | 0 |
| 3.5 | 1 | 3.5 |
| 3.6 | 2 | 7.2 |
| 3.7 | 1 | 3.7 |
| 3.8 | 3 | 11.4 |
| 3.9 | 3 | 11.7 |
| 4.0 | 4 | 16.0 |
| 4.1 | 3 | 12.3 |
| 4.2 | 2 | 8.4 |
| 4.3 | 2 | 8.6 |
| 4.4 | 1 | 4.4 |
| 4.5 | 1 | 4.5 |
| Sum | 24 | 95 |
24개의 관측치 중 11개의 관측치는 4.0cm 보다 작고 9개는 크다.
그러므로 통상적인 중위수는 12번째 값이 포함된 4.0 이라 하기는 어렵다.
하지만 중위수는 [3.95,4.05] 이내에 있을 필요가 있다고 생각해야한다.
따라서 예상구간의 하한값은 3.95 가 된다.
2) 총 빈도
sum(fi)
## [1] 24
누적빈도는 4.0이 오기 전 까지의 누적 빈도를 사용한다.
which(fi==4.0) # 8번째 인덱스에 4.0 위치
## [1] 8
cf <- cumsum(fi)
cf[7] #7번째 인덱스 까지의 누적빈도
## [1] 11
diff(xi) #0.1
## [1] 0.1 0.1 0.1 0.1 0.1 0.1 0.1 0.1 0.1 0.1 0.1 0.1
library('ggplot2')
ggplot(ex3_1, aes(x=Length)) + geom_histogram(binwidth = 0.1, fill='#8dd3c7',colour='#ffffb3')+
scale_x_continuous(breaks = seq(0,4.5,0.1))
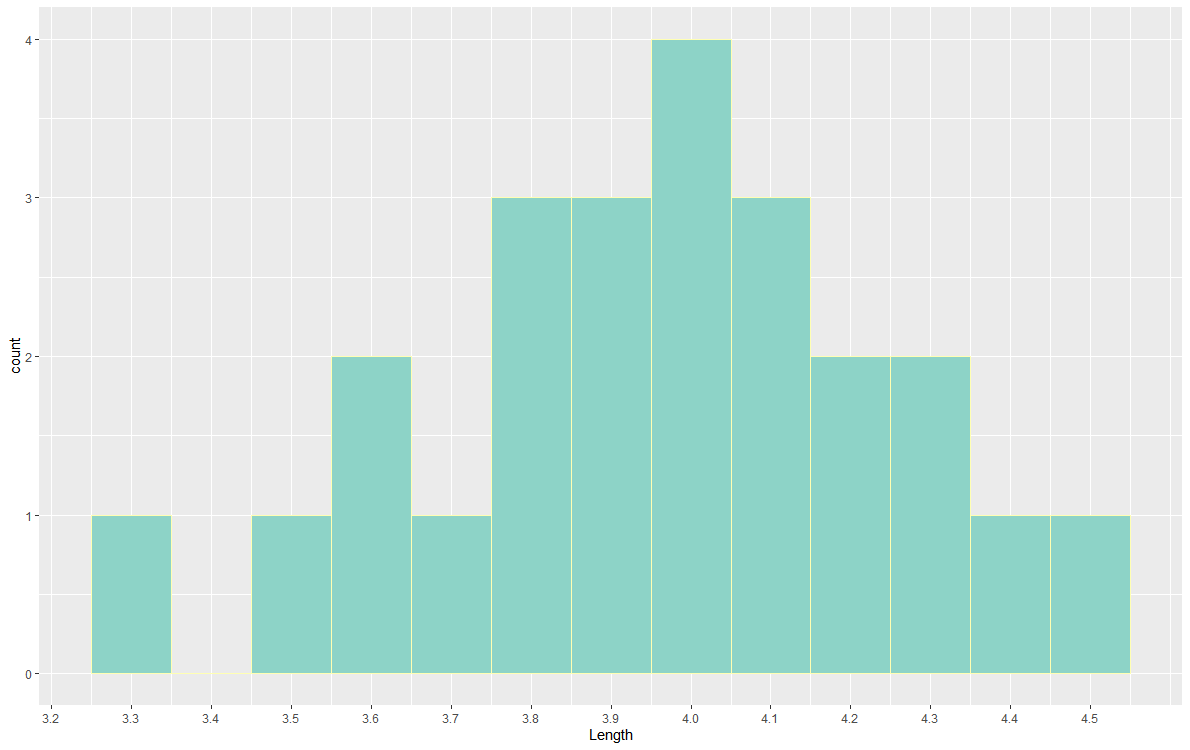
- 히스토그램
- 히스토그램을 보면 날개의 길이가 4인 경우의 빈도수가 4로 가장 많은것을 알 수 있다.
- 나비 날개 길이의 frequency table 출력 결과를 살펴보면
표본평균 값이 3.96이고 중앙값이 3.975이므로 두 값이 서로 비슷한 것을 확인할 수 있다.
따라서 거의 치우치지 않은 분포의 형태를 띄고 있는 것을 알 수 있고 히스토그램을 통해서도 확인할 수 있다.
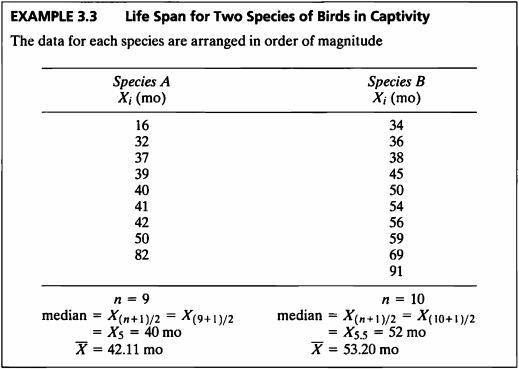
EXAMPLE 3.3
#데이터셋
ex3_3
## exam3_3.Group exam3_3.LifeSpan
## 1 A 16
## 2 A 32
## 3 A 37
## 4 A 39
## 5 A 40
## 6 A 41
## 7 A 42
## 8 A 50
## 9 A 82
## 10 B 34
## 11 B 36
## 12 B 38
## 13 B 45
## 14 B 50
## 15 B 54
## 16 B 56
## 17 B 59
## 18 B 69
## 19 B 91
ex3_3$exam3_3.LifeSpan
## [1] 16 32 37 39 40 41 42 50 82 34 36 38 45 50 54 56 59 69 91
ex3_3_a <- subset(ex3_3,exam3_3.Group=="A")
ex3_3_b <- subset(ex3_3,exam3_3.Group=="B")
a_n <- length(ex3_3_a$exam3_3.Group)
b_n <- length(ex3_3_b$exam3_3.Group)
a_med <- median(ex3_3_a$exam3_3.LifeSpan)
b_med <- median(ex3_3_b$exam3_3.LifeSpan)
a_mn <- round(mean(ex3_3_a$exam3_3.LifeSpan),2) ;
b_mn <- mean(ex3_3_b$exam3_3.LifeSpan)
cat(" Number of Species A = ",a_n,", Median of Species A = ",a_med, ", Mean of Species A = ",a_mn,"\n",
"Number of Species B = ",b_n,", Median of Species B = ",b_med, ", Mean of Species B = ",b_mn)
## Number of Species A = 9 , Median of Species A = 40 , Mean of Species A = 42.11
## Number of Species B = 10 , Median of Species B = 52 , Mean of Species B = 53.2
| Species A | Species B | |
|---|---|---|
| \(n\) | 9 | 10 |
| \(median\) | 40 | 52 |
| \(\overline X\) | 42.11 | 53.20 |
EXAMPLE 3.4

#데이터셋
ex3_4
## exam3_4.decade exam3_4.population exam3_4.ratio
## 1 0 10000 NA
## 2 1 10500 1.05
## 3 2 11550 1.10
## 4 3 13860 1.20
## 5 4 18156 1.31
- Manually
x_bar <- mean(na.omit(ex3_4$exam3_4.ratio))
geo_mean_x_method1 <- exp(mean(log(na.omit(ex3_4$exam3_4.ratio))))
geo_mean_x_method2 <- prod(na.omit(ex3_4$exam3_4.ratio)) ^ (1/length(na.omit(ex3_4$exam3_4.ratio)))
cat('Geometric Mean Manually =', '(' ,'method1 =', geo_mean_x_method1, ',' ,'method2 =' ,geo_mean_x_method2,')')
## Geometric Mean Manually = ( method1 = 1.160803 , method2 = 1.160803 )
- Package
library(psych)
geo_mean_x_package <- geometric.mean(ex3_4$exam3_4.ratio) ; cat('Geometric Mean with psych package =', geo_mean_x_package)
## Geometric Mean with psych package = 1.160803
데이터 로그 변환의 산술 평균을 다시 역로그변환(지수변환) (Method 2)으로 나타낼 수 있다.
변화율의 산술 평균은 1.165이며, 이 때 기하 평균은 두 가지 방법 및 패키지를 이용하였을 때 1.1608로 동일하게 산출된다.
first_p <- ex3_4$exam3_4.population[1] # 초기 population
final_p_a <- round(first_p*mean(ex3_4$exam3_4.ratio[2:5])^4)
final_p_g <- round(first_p*round(geometric.mean(ex3_4$exam3_4.ratio[2:5]),4)^4)
cat(" 산술평균으로 구한 Final Population = ",final_p_a,"\n","기하평균으로 구한 Final Population = ",final_p_g)
## 산술평균으로 구한 Final Population = 18421
## 기하평균으로 구한 Final Population = 18156
EXAMPLE 3.5

#데이터셋
ex3_5
## X
## 1 40
## 2 20
- Manually
harmonic_mean_manual <- round(length(ex3_5$X) / sum(1/ex3_5$X),2) ; cat('Harmonic Mean Manually =', harmonic_mean_manual)
## Harmonic Mean Manually = 26.67
- Package
library(psych)
harmonic_mean_package <- round(harmonic.mean(ex3_5$X),2) ; cat('Harmonic Mean with psych package =', harmonic_mean_package)
## Harmonic Mean with psych package = 26.67
xm <- mean(ex3_5$X)
cat("산술평균=", xm, "km/h, 조화평균=",harmonic_mean_manual,"km/h")
## 산술평균= 30 km/h, 조화평균= 26.67 km/h
평균속력은 40과 20의 조화평균인 26.67 km/h가 된다.
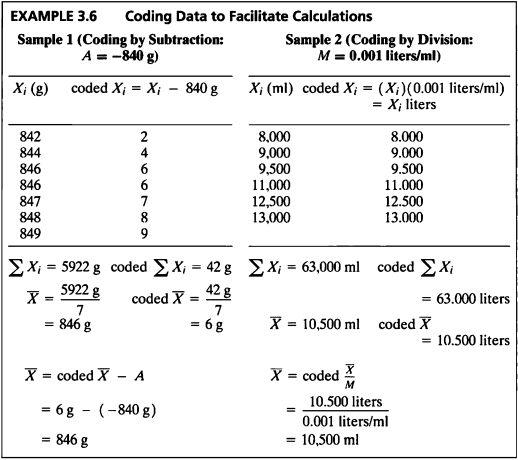
EXAMPLE 3.6
#데이터셋
ex3_6
## exam3_6.X1 exam3_6.code1 exam3_6.X2 exam3_6.code2
## 1 842 2 8000 8000
## 2 844 4 9000 9000
## 3 846 6 95000 95000
## 4 846 6 11000 11000
## 5 847 7 12500 12500
## 6 848 8 13000 13000
## 7 849 9 NA NA
The mean of the coded values is equal to \(\overline X - 840 g\).
A <- -840 ; M <- 0.001
sum_sample1 <- sum(ex3_6$exam3_6.X1)
coded_sum_sample1 <- sum(ex3_6$exam3_6.code1)
mean_sample1 <- mean(ex3_6$exam3_6.X1)
coded_mean_sample1 <- mean(ex3_6$exam3_6.code1)
mean_sample1 <- coded_mean_sample1-A
mean_sample1
## [1] 846
The mean of the coded values is equal to \(\overline X * M\).
#데이터 오기 수정하기
ex3_6$exam3_6.X2[3] <- ex3_6$exam3_6.X2[3]/10
ex3_6$exam3_6.code2[3] <- ex3_6$exam3_6.code2[3]/10
#결측값제외하고 계산
sum_sample2 <- sum(na.omit(ex3_6$exam3_6.X2))
coded_sum_sample2 <- sum(na.omit(ex3_6$exam3_6.code2*M))
mean_sample2 <- mean(na.omit(ex3_6$exam3_6.X2))
coded_mean_sample2 <- mean(na.omit(ex3_6$exam3_6.code2*M))
mean_sample2 <- coded_mean_sample2 / M
mean_sample2
## [1] 10500
| stat / Group | Sample 1 | Sample 2 |
|---|---|---|
| Sum | 5922 | 63000 |
| coded Sum | 42 | 63 |
| Mean | 846 | 10500 |
| coded Mean | 6 | 10.5 |
SAS 프로그램 결과
sas 접기/펼치기 버튼
3장
LIBNAME ex 'C:\Biostat';
RUN;
/*3장 연습문제 불러오기*/
%macro chap03(name=,no=);
%do i=1 %to &no.;
PROC IMPORT DBMS=excel
DATAFILE="C:\Biostat\data_chap03"
OUT=ex.&name.&i. REPLACE;
RANGE="exam3_&i.$";
RUN;
%end;
%mend;
%chap03(name=ex3_,no=6);
EXAMPLE 3.1
PROC MEANS DATA=ex.ex3_1 NOPRINT;
VAR length;
OUTPUT OUT=ex.ex3_1stat(drop=_type_ _freq_) sum=sum n=sample_size mean=mean;
run;
PROC PRINT DATA=ex.ex3_1stat NOOBS;
RUN;
| sum | sample_size | mean |
|---|---|---|
| 95 | 24 | 3.95833 |
EXAMPLE 3.2
/*빈도 곱한 데이터 생성*/
DATA ex.ex3_2_sum;
SET ex.ex3_2 ;
fX = X * Freq;
RUN;
PROC IML;
USE ex.ex3_2_sum;
READ all;
CLOSE exam3_2_sum;
k = nrow(X);
n = sum(Freq); /*총빈도*/
n_divided_by_2 = n/2;
X_bar = round(sum(fX) / n, 0.01);
L = (3.9+4.0)/2; /*예상구간의 하한값*/
G=Freq[8]; /*예상구간빈도*/
B=Freq[1]+Freq[2]+Freq[3]+Freq[4]+Freq[5]+Freq[6]+Freq[7]; /*누적빈도*/
W = X[2] - X[1];
Estimated_median = L + ((n/2)-B)/G * W;
PRINT k n X_bar n_divided_by_2 L G B W Estimated_median ;
RUN;
PROC SGPLOT DATA=ex.ex3_2;
histogram x / group=freq scale=count freq=freq;
xaxis label='Wing Length in cm' grid values= (3.3 to 4.5 by 0.1);
yaxis label='Frequency' grid values= (0 to 4 by 1);
RUN;
| k | n | X_bar | n_divided_by_2 | L | G | B | W | Estimated_median |
|---|---|---|---|---|---|---|---|---|
| 13 | 24 | 3.96 | 12 | 3.95 | 4 | 11 | 0.1 | 3.975 |
EXAMPLE 3.3
PROC MEANS DATA=ex.ex3_3 MEAN MEDIAN MAXDEC=2;
CLASS group;
VAR lifespan;
RUN;
MEANS 프로시저
| 분석 변수: LifeSpan LifeSpan | |||
|---|---|---|---|
| Group | 관측값 수 | 평균 | 중위수 |
| A | 9 | 42.11 | 40.00 |
| B | 10 | 53.20 | 52.00 |
EXAMPLE 3.4
/*GM*/
PROC SURVEYMEANS DATA=ex.ex3_4 MEAN GEOMEAN PLOTS=none;
VAR ratio;
RUN;
DATA ex.ex3_4_keep;
SET ex.ex3_4;
KEEP ratio;
IF ratio=. THEN DELETE;
RUN;
PROC IML;
USE ex.ex3_4_keep;
READ all;
CLOSE ex.ex3_4_keep;
arithmetic_mean = round( mean(ratio), 0.0001) ;
Final_population1 = round(10000*(arithmetic_mean**4), 1);
geometric_mean = round( geomean(ratio), 0.0001);
Final_population2 = round(10000*(geometric_mean**4), 1);
PRINT arithmetic_mean Final_population1 geometric_mean Final_population2;
TITLE 'The Geometric Mean of Ratios of Changes';
RUN;
QUIT;
The SURVEYMEANS Procedure
| Data Summary | |
|---|---|
| Number of Observations | 5 |
| Statistics | |||
|---|---|---|---|
| Variable | Label | Mean | Std Error of Mean |
| ratio | ratio | 1.165000 | 0.057518 |
| Geometric Means | |||
|---|---|---|---|
| Variable | Label | Geometric Mean | Std Error |
| ratio | ratio | 1.160803 | 0.056751 |
| arithmetic_mean | Final_population1 | geometric_mean | Final_population2 |
|---|---|---|---|
| 1.165 | 18421 | 1.1608 | 18156 |
EXAMPLE 3.5
PROC IML;
USE ex.ex3_5;
READ all;
CLOSE ex.ex3_5;
mean=mean(x);
har_mean= round(2/(sum(1/x)),0.01);
print mean har_mean;
RUN;
QUIT;
| mean | har_mean |
|---|---|
| 30 | 26.67 |
EXAMPLE 3.6
PROC IML;
use ex.ex3_6; read all; close ex.ex3_6;
A=840;
mean1=mean(x1);
mean2 = mean(code1);
raw_mean = mean2 +A;
print mean1 mean2 raw_mean;
RUN;
QUIT;
DATA ex.ex3_6_1;
set ex.ex3_6;
if x2='.' then delete;
if code2='.' then delete;
if x2=95000 then x2=x2/10;
RUN;
PROC IML;
use ex.ex3_6_1; read all; close ex.ex3_6_1;
B=0.001;
B2= x2*B;
mean1= mean(x2);
mean2= mean(B2);
raw_mean=mean2/B;
print mean1 mean2 raw_mean;
RUN;
QUIT;
| mean1 | mean2 | raw_mean |
|---|---|---|
| 846 | 6 | 846 |
| mean1 | mean2 | raw_mean |
|---|---|---|
| 10500 | 10.5 | 10500 |
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
