[의학통계방법론] Ch1. Data Types and Presentation
Data Types and Presentation
정리내용 접기/펼치기 버튼
1.1 의생물 데이터의 유형
- 자료
- 양적 자료: 이산형, 연속형
- 질적 자료: 명목형, 순서형
- 측정 (measurement) 이란?
(일정한 규칙에 의해 대상, 사건, 상태의 특성에 숫자를 부여하는 것)- 위치 측도 (measures of location)
- 산포 측도 (measures of spread)
- 신뢰도 측도 (measures of reliability)
- 측정 척도(scale of measure; level of measurement)
(일정한 규칙에 의해 부여된 수치들의 내재적 특성을 설명하기 위한 분류)- 질적 척도: 명목척도, 서열척도(=순위척도)
- 양적 척도: 구간척도(=등간척도), 비척도
- 질적 데이터 (qualitative data): 크기나 양적 차이 구분이 불가능
- 명목척도(nominal scale): 속성의 유무만을 나타내며 양적 크기는 못 나타낸다.
- 생사여부, 성별, 치료 여부
- 질병 상태, 결혼 상태
- 혈액형
- 직종
- 서열척도 (ordinal scale): 만족도, 행복 정도, 불쾌 지수 같이 비수치형으로 순서성만 나타낸다.
- 중증도
- 출생서열: 첫째, 둘째, 셋째 등
- 5정 리커트 척도
- 암의 병기(stage): I, II, III, 또는 IV
- 통계분석을 위해 질적 데이터는 통상적으로 서로 다른 값으로 코딩 한 후 빈도 측정함으로써 이산형 숫자로 변환된다.
- 명목척도(nominal scale): 속성의 유무만을 나타내며 양적 크기는 못 나타낸다.
- 양적 데이터 (quantitative data): 크기나 양적 차이 구분이 가능
- 구간척도(interval scale): 구간척도(=등간척도)는 절대 O(true zero)이 없음. 간격이 일정함.
- 체온
- 각종 지수(index)
- 일,월,년
- 비척도(ratio scale): 비척도는 절대 0이 있으며 크기의 비교가 가능하여 수의 사칙연산 적용 가능함.
- 이산형 (numerical discrete)
- 임신 회수(parity),사망자 수(number of deaths)
- 매년 한국에서 AIDS에 걸리는 새로운 환자 수(counts)
- 자녀 수
- 연속형 (numerical continuous)
- 체중, 결혼 연령
- 생존시간
- 혈압
- 중량, 부피, 용량, 비율, 길이 등
- 이산형 (numerical discrete)
- 구간척도(interval scale): 구간척도(=등간척도)는 절대 O(true zero)이 없음. 간격이 일정함.
- 보충
- 비 (ratio)
- 측정 단위가 같은 두 개 값 간의 상대적 크기를 비교한 것 (예: A:B)
- 분자와 분모가 서로 독립적인 관계로 서로 다른 범주일 때 사용
- A에 대한 B의 비는 B/A로 계산 됨.
예) 성비, 인구밀도(인구/면적) 등이 비에 해당됨
남자:여자 = 60:40 (명)
- 비율 (proportion)
- 전체를 1로 보았을 때 한 항목이 차지하는 값
- 비의 특수한 형태로 분모에 분자가 포함됨.
예) 전체 미혼자 중 남자 미혼자의 비율
남자 미혼자/(남자 미혼자 + 여자 미혼자).
- 백분율 (percent)
- 전체를 100으로 보았을 때 한 항목이 차지하는 값
- 백분율 = 비율 x 100
- 율 (rate)
- 모집단의 구성원 수 중 사건의 수로 표현함
- 서로 측정 단위가 다른 두 수치 간의 비 (ratio) 임.
- 비 (ratio)
1.2 Accuracy and Significant Figures
- 정확도와 정밀도 (accuracy and precision)
- 정확도 (accuracy)
- 측정되는 변수의 참값에 가깝게 측정되고 있는 정도를 나타낸 것.
The nearness of a measurement to the true value of the variable being measured.
(측정의 정확성)
- 측정되는 변수의 참값에 가깝게 측정되고 있는 정도를 나타낸 것.
- 정밀도 (precision)
- 같은 양을 반복측정할 때 매번의 측정값들이 비슷하게 측정되고 있는 정도를 나타낸 것.
It’s not a synonymous term but refers to the closeness to each other of repeated measurements of the same quantity (측정의 재현성)
- 같은 양을 반복측정할 때 매번의 측정값들이 비슷하게 측정되고 있는 정도를 나타낸 것.
- 정확도 (accuracy)
1.3 Frequency Distributions
- 연속형 자료를 범주화 할 땐 범주를 명확하게 표시해야한다.
- 빈도분포를 살펴봄으로 이질적인 자료가 모인 것은 아닌지 예상한다.
이봉분포시 이질적인 자료가 모인 것으로 예상한다.
ex) 수술자가 다르다면 수술자 정보를 알아야 한다.
1.4 Cumulative Frequency Dstributions
- Cumulative Frequency Dstributions는 중위수, 백분위수 및 기타 분위수를 결정하는데 유용하다.
- 데이터를 살펴볼 때
- 데이터 분포의 모양, 형태를 살피기
- 데이터의 중심 위치파악
- 명목척도일 경우 최빈값을 이용
- 분포가 대칭이고 극단값이 없는 경우 산술평균이나 중위수 모두 이용
- 분포가 비대칭이고 극단값이 있는 경우 중위수를 대표값으로 선택하고 산술평균을 보조적으로 제시
- 순위척도일 경우 중위수를 대표값으로 이용
- 데이터의 범위파악
- 분포의 형태는 어떠한가?
예: 정규분포, 이봉 분포(bimodal distribution), 지수분포, 치우친분포(skewed distribution) 등.
대칭/비대칭 - 왜도
뾰족한가/완만한가 - 첨도 - 이상치 (outlier)들이 있는가?
- 데이터 값들 중 비상식적으로 이상한 값이 존재하는가?
- 정보의 약 90%는 그래프에 포함되어 있다고 한다.
- 통계학의 첫째 규칙 : 상식을 이용하기
R 프로그램 결과
R 접기/펼치기 버튼
패키지 설치된 패키지 접기/펼치기 버튼
getwd()
## [1] "C:/Biostat"
library("readxl")
library("ggplot2")
library("showtext")
엑셀파일불러오기
library('readxl')
#모든 시트를 하나의 리스트로 불러오는 함수
read_excel_allsheets <- function(file, tibble = FALSE) {
sheets <- readxl::excel_sheets(file)
x <- lapply(sheets, function(X) readxl::read_excel(file, sheet = X))
if(!tibble) x <- lapply(x, as.data.frame)
names(x) <- sheets
x
}
1장
1장 연습문제 불러오기
#data_chap01에 연습문제 1장 모든 문제 저장
data_chap01 <- read_excel_allsheets("data_chap01.xls")
#연습문제 각각 데이터 생성
for (x in 1:length(data_chap01)){
assign(paste0('ex1_',1:length(data_chap01))[x],data_chap01[x])
}
#연습문제 데이터 형식을 리스트에서 데이터프레임으로 변환
for (x in 1:length(data_chap01)){
assign(paste0('ex1_',1:length(data_chap01))[x],data.frame(data_chap01[x]))
}
EXAMPLE 1.1

위치에 따른 새 둥지의 갯수
#데이터셋
ex1_1
## exam1_1.NestSite exam1_1.Number
## 1 A. Vines 56
## 2 B. Building caves 60
## 3 C. Low tree branches 46
## 4 D. Tree and building cavities 49
library('ggplot2')
#둥지 개수를 bar graph로 출력하기
ggplot(ex1_1) +
aes(x = exam1_1.NestSite, weight = exam1_1.Number) +
scale_y_continuous(breaks = seq(0,60,10),limits=c(0,60))+
geom_bar(fill = c("#8dd3c7","#ffffb3","#bebada","#fb8072")) +
labs(y = "Number of Nests",
title = "The Location of Sparrow Nest",
subtitle = ": Frequency Table of Nominal Data") +
theme_minimal()
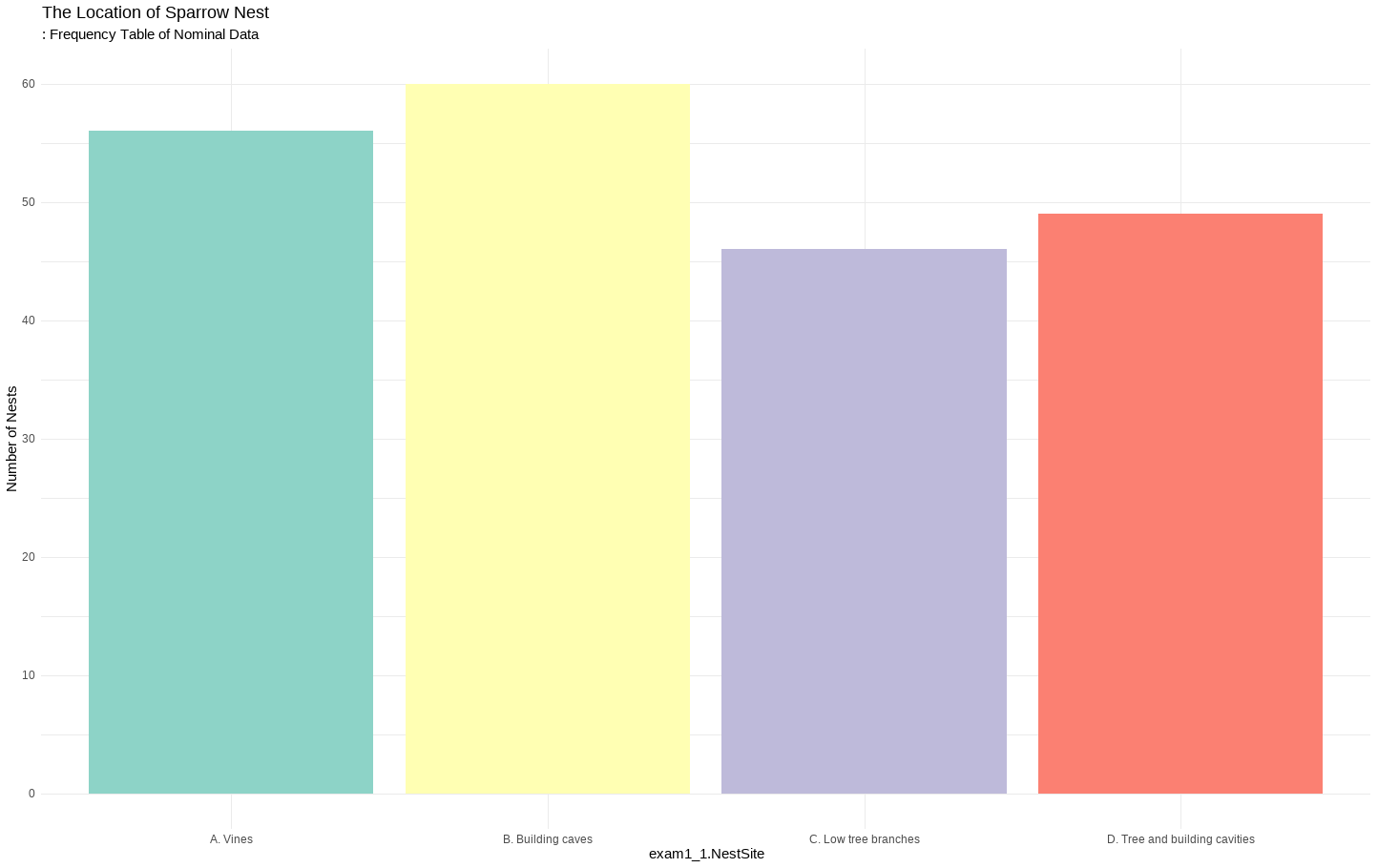
- 첫번째 막대그래프를 통해 참새 둥지의 위치 빈도를 살펴본 결과 건물 처마에서 가장 높은 빈도를 보였으며 낮은 나무의 나뭇가지에서 가장 낮은 빈도를 보였다.
- 앞서 살펴본 그래프의 경우 각 위치에 대해 빈도의 범위가 0부터 60이었기 때문에 각 위치별로 빈도의 차이가 크게 나타나지 않았다.
#글씨체를 바꾸기 위해 폰트를 다운받는다.
library(showtext)
font_add_google("Raleway", family="raleway")
font_add_google("Montserrat", family="mont")
showtext_auto()
library(ggplot2)
library(grid)
bar1 <- ggplot(ex1_1, aes(x=exam1_1.NestSite, y=exam1_1.Number,fill=exam1_1.NestSite))+
geom_bar(stat="identity")+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.1 Bar graph of Sparrow Nest")+
geom_text(aes(label=exam1_1.Number),vjust=-0.3,size=8)+
geom_text(aes(label=paste0(round((exam1_1.Number/sum(exam1_1.Number))*100),"%")),vjust=5,size=8,family = "mont")+
scale_fill_brewer(palette="Set3")+
ylab("Number of Nests Observed")+
theme_bw()+
theme(legend.position = "bottom")+
theme(legend.text = element_text(size=20))+
theme(legend.title=element_blank())+
theme(axis.title=element_text(size=20),title = element_text(size=30))+
theme(axis.text.x=element_text(size=15))+
annotate("text", x=3.15, y=58, label=paste0("Total = ",round(sum(ex1_1$exam1_1.Number),2)),family="mont", size=10,hjust=0)+
theme()
bar1
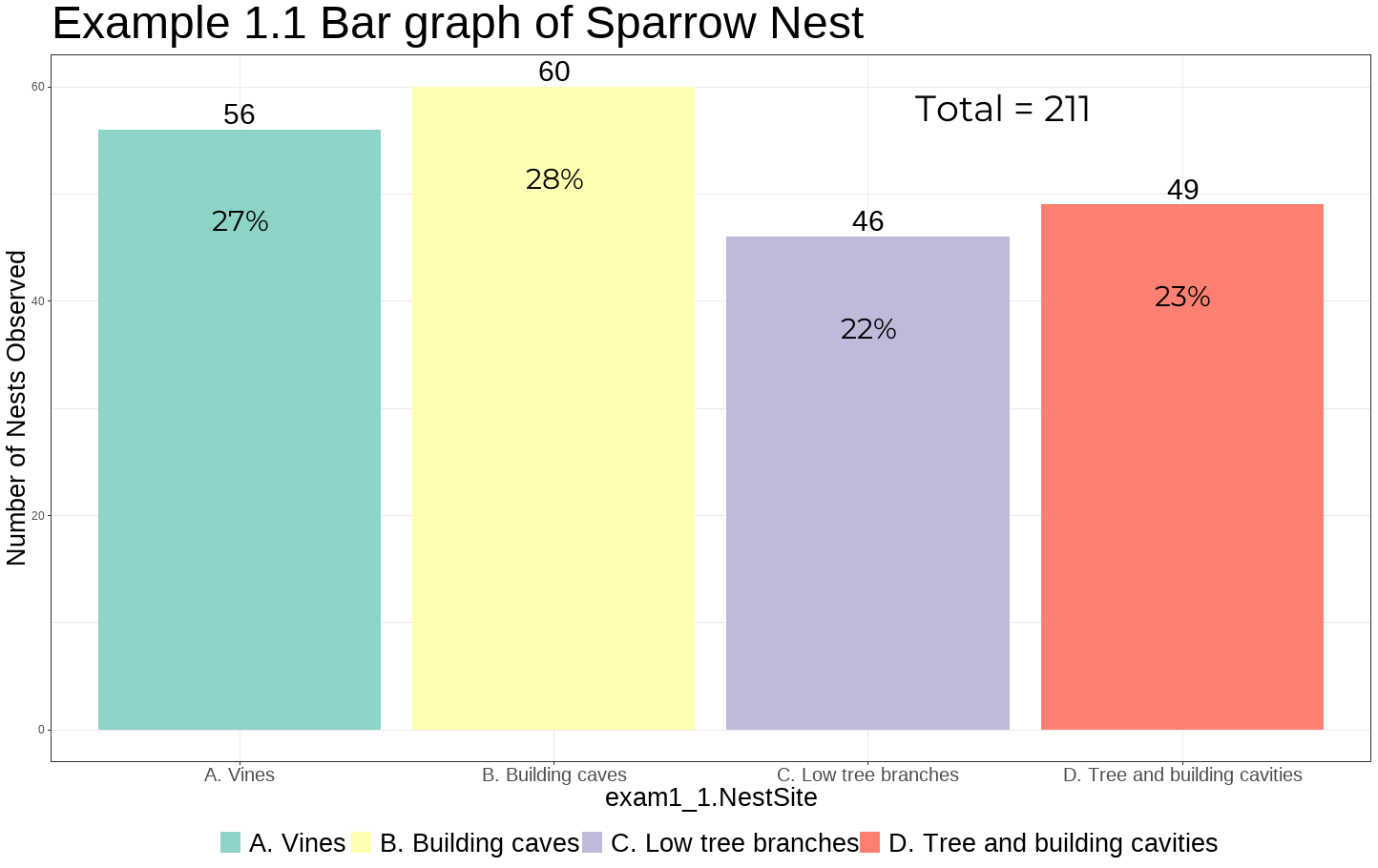
- 그래프를 보면 관찰된 참새들의 둥지 수는 총 211개이며 그 중 가장 많이 관측된 곳은 B, 즉 빌딩에 있는 굴이며 총 60개가 관측되었으며 이는 관측된 둥지의 수 중 약 28%를 차지하고 있으며 가장 작은 비율을 가진 곳은 C이며 낮은 나무의 나뭇가지로 46개가 관측되었다.
bar2 <- ggplot(ex1_1, aes(x=exam1_1.NestSite, y=exam1_1.Number,fill=exam1_1.NestSite))+
geom_bar(stat="identity")+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.1 Bar graph of Sparrow Nest (over 45)")+
geom_text(aes(label=exam1_1.Number-45),vjust=-0.3,size=8)+
geom_text(aes(label=paste0(round(((exam1_1.Number-45)/sum(exam1_1.Number-45))*100),"%")),vjust=3,size=8,family = "raleway")+
scale_fill_brewer(palette="Set3")+
ylab("Number of Nests Observed")+
theme_bw()+
theme(legend.position = "bottom")+
theme(legend.text = element_text(size=20))+
theme(legend.title=element_blank())+
theme(axis.title=element_text(size=20),title = element_text(size=30))+
theme(axis.text.x=element_text(size=15))+
theme(axis.text.y=element_text(size=15))+
annotate("text", x=3.15, y=58, label=paste0("Total = ",round(sum(ex1_1$exam1_1.Number -45),2)),family="mont", size=10,hjust=0)+
coord_cartesian(ylim=c(45,60))+
theme()
bar2
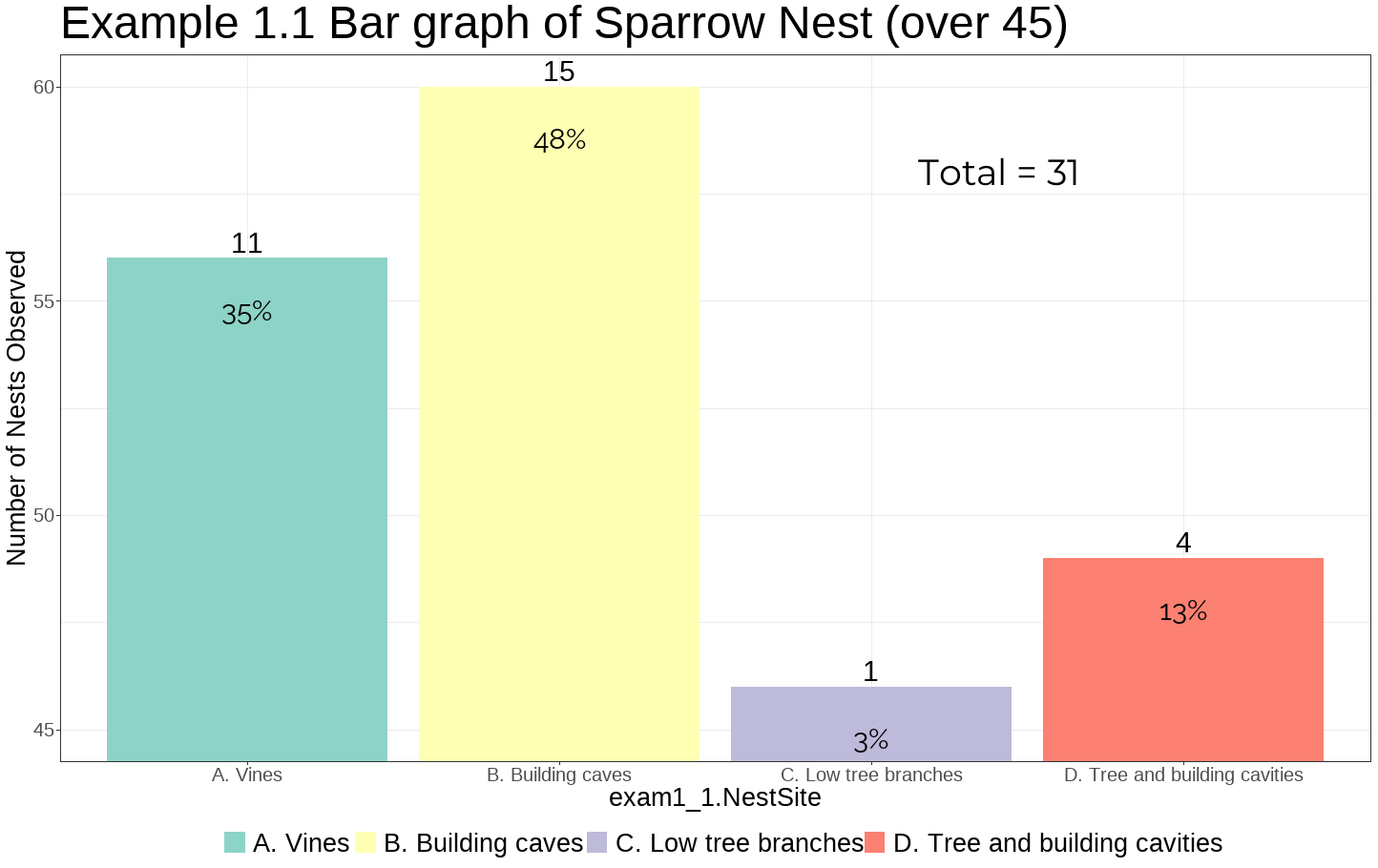
- 반면 빈도의 범위를 45부터 60으로 수정하여 그래프를 작성한 결과
다음과 같이 각 위치별 빈도가 눈에 띄게 차이를 보이는 것을 알 수 있다.
따라서 이 예제를 보아 시각화의 중요성을 볼 수 있다.
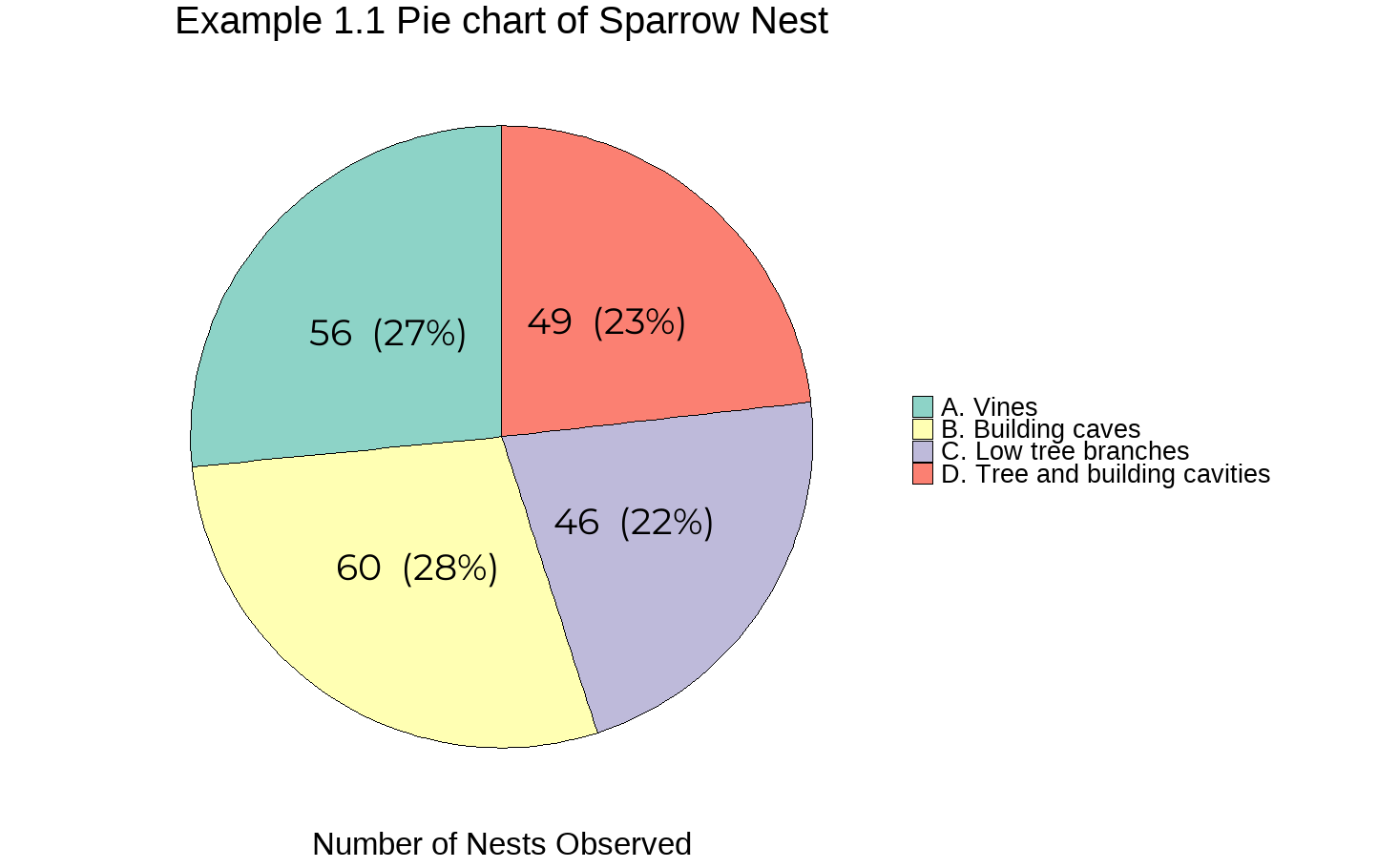
Example 1.1 의 자료를 파이차트로 그리면 다음과 같다.
pie1<- ggplot(ex1_1, aes(x='', y=ex1_1$exam1_1.Number,fill=ex1_1$exam1_1.NestSite))+
geom_bar(stat="identity",colour="black")+
coord_polar('y')+
theme(plot.title = element_text(size=30,hjust = 0.9))+
ggtitle("Example 1.1 Pie chart of Sparrow Nest")+
geom_text(aes(label=paste0(ex1_1$exam1_1.Number," (",round((ex1_1$exam1_1.Number/sum(ex1_1$exam1_1.Number))*100),"%)"),family = "mont"),
position=position_stack(vjust=0.5),size=10,fontface="plain")+
scale_fill_brewer(palette="Set3")+
ylab("Number of Nests Observed")+
labs(x=NULL,fill=NULL)+
theme_classic()+
theme(axis.line=element_blank(),
axis.text = element_blank(),
axis.ticks = element_blank(),
plot.title = element_text(size=30,hjust=0.5),
axis.title = element_text(size=25),
legend.text = element_text(size=20))
pie1
EXAMPLE 1.2

#데이터셋
ex1_2
## exam1_2.Class exam1_2.Amount exam1_2.Number
## 1 0 No black pigmentation 13
## 2 1 Faintly speckled 68
## 3 2 Moderately speckled 44
## 4 3 Heavily speckled 21
## 5 4 Solid black pigmentation 8
- 위의 데이터는 개복치와 색소침착에 대한 데이터이다.
이를 막대 그래프로 표현하면 다음과 같다.
bar1_2 <- ggplot(ex1_2, aes(x=exam1_2.Class, y=exam1_2.Number,fill=exam1_2.Amount))+
geom_bar(stat="identity")+
ggtitle("Example 1.2 Bar plot of Sunfish pigmentation")+
geom_text(aes(label=exam1_2.Number),vjust=-0.3,size=10)+
geom_text(aes(label=paste0(round((exam1_2.Number/sum(exam1_2.Number))*100),"%")),vjust=2,size=10,family = "mont")+
scale_fill_brewer(palette="Set3")+
labs(fill="Amount of Pigmentation")+
xlab("Pigmentation Class")+
ylab("Number of Fish")+
theme_bw()+
theme(legend.position = "right")+
theme(legend.text = element_text(size=20))+
theme(legend.title = element_text(size=20))+
theme(axis.text.x = element_text(size=20))+
theme(axis.text.y=element_text(size=20))+
theme(axis.title = element_text(size=20))+
theme(plot.title = element_text(size=30,hjust = 0.5))+
annotate("text", x=2.5, y=58, label=paste0("Total = ",round(sum(ex1_2$exam1_2.Number),2)),family="mont", size=10,hjust=0)
bar1_2
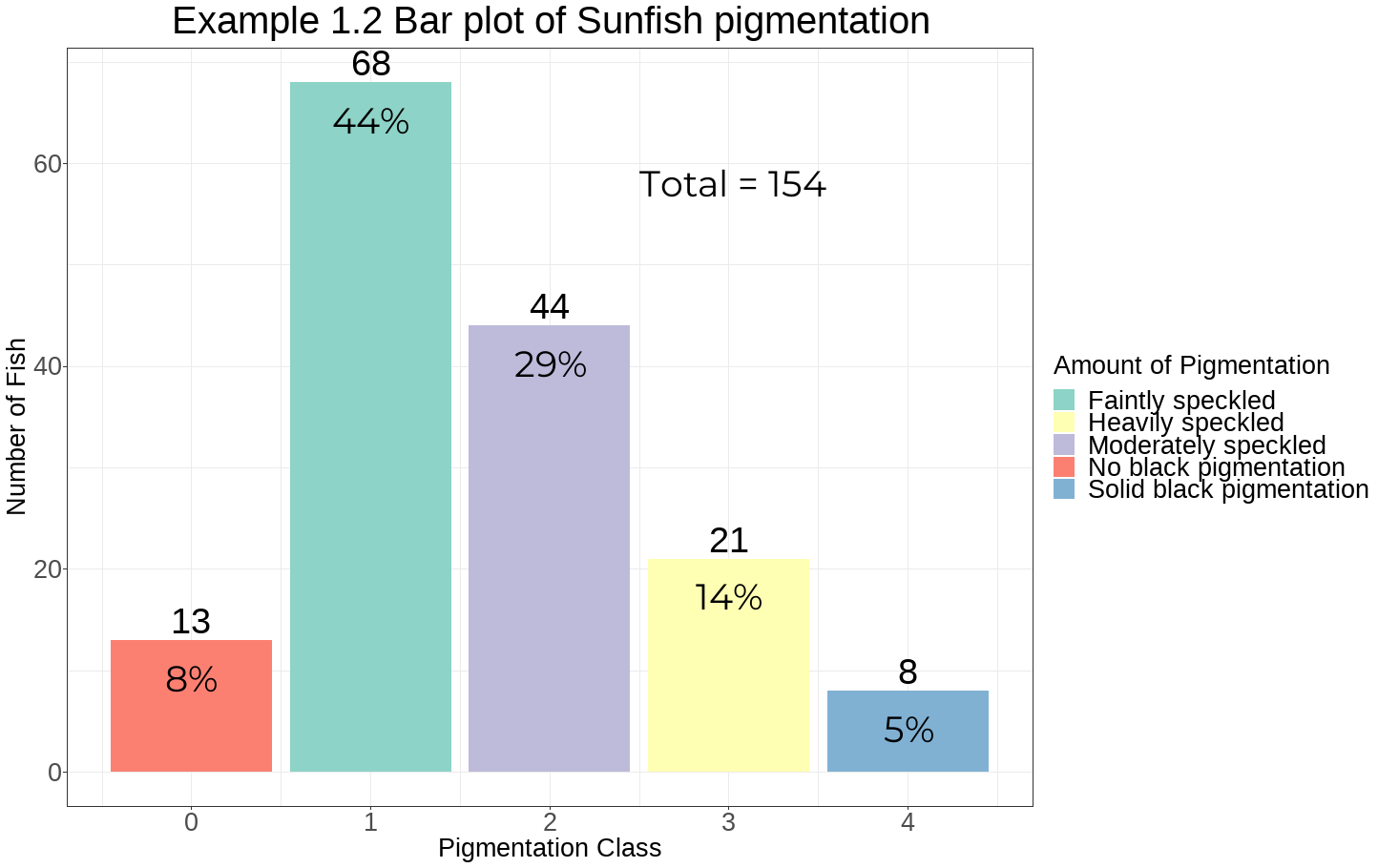
- 검정색 색소 양에 따른 개복치의 개체수를 그래프로 나타낸 결과 희미하게 얼룩덜룩한 정도의 개복치 개체수가 가장 많았고 중간정도, 심하게 얼룩덜룩한 정도가 그 뒤를 이었다.
- 완전히 검정색인 개체수가 가장 적게 나타났으며 아예 검정색 색소가 없는 개체수 또한 매우 적게 나타났다.
- 총 개복치의 수는 154 마리이고 그 중 Faintly speckled(희미하게 얼룩덜룩한 정도의 개복치)의 수가 65로 전체 중 약 44%를 차지하고 있음을 알 수 있다.
EXAMPLE 1.3

#데이터셋
ex1_3
## exam1_3.Size exam1_3.Frequency
## 1 3 10
## 2 4 27
## 3 5 22
## 4 6 4
## 5 7 1
- 여우의 한배새끼 수를 나타낸 데이터이다.
한배새끼 수란 1회 분만할 때 출산하는 새끼의 수를 말한다.
bar1_3 <- ggplot(ex1_3, aes(x=exam1_3.Size, y=exam1_3.Frequency,fill=c("3","4","5","6","7")))+
geom_bar(stat="identity")+
ggtitle("Example 1.3 Bar plot of Litter size of Foxes")+
geom_text(aes(label=exam1_3.Frequency),vjust=-0.3,size=10)+
geom_text(aes(label=paste0(round((exam1_3.Frequency/sum(exam1_3.Frequency))*100),"%")),vjust=1.5,size=6,family = "mont")+
scale_fill_brewer(palette="Set3")+
labs(fill="Amount of Pigmentation")+
xlab("Litter Size")+
ylab("Number of Litters")+
theme_bw()+
theme(legend.position = "right")+
theme(legend.text = element_text(size=20))+
theme(legend.title = element_text(size=20))+
theme(axis.text.x = element_text(size=20))+
theme(axis.text.y = element_text(size=20))+
theme(axis.title = element_text(size=20))+
theme(plot.title = element_text(size=30,hjust = 0.5))+
annotate("text", x=2.5, y=30, label=paste0("Total = ",round(sum(ex1_3$exam1_3.Frequency),2)),family="mont", size=10,hjust=0)
bar1_3
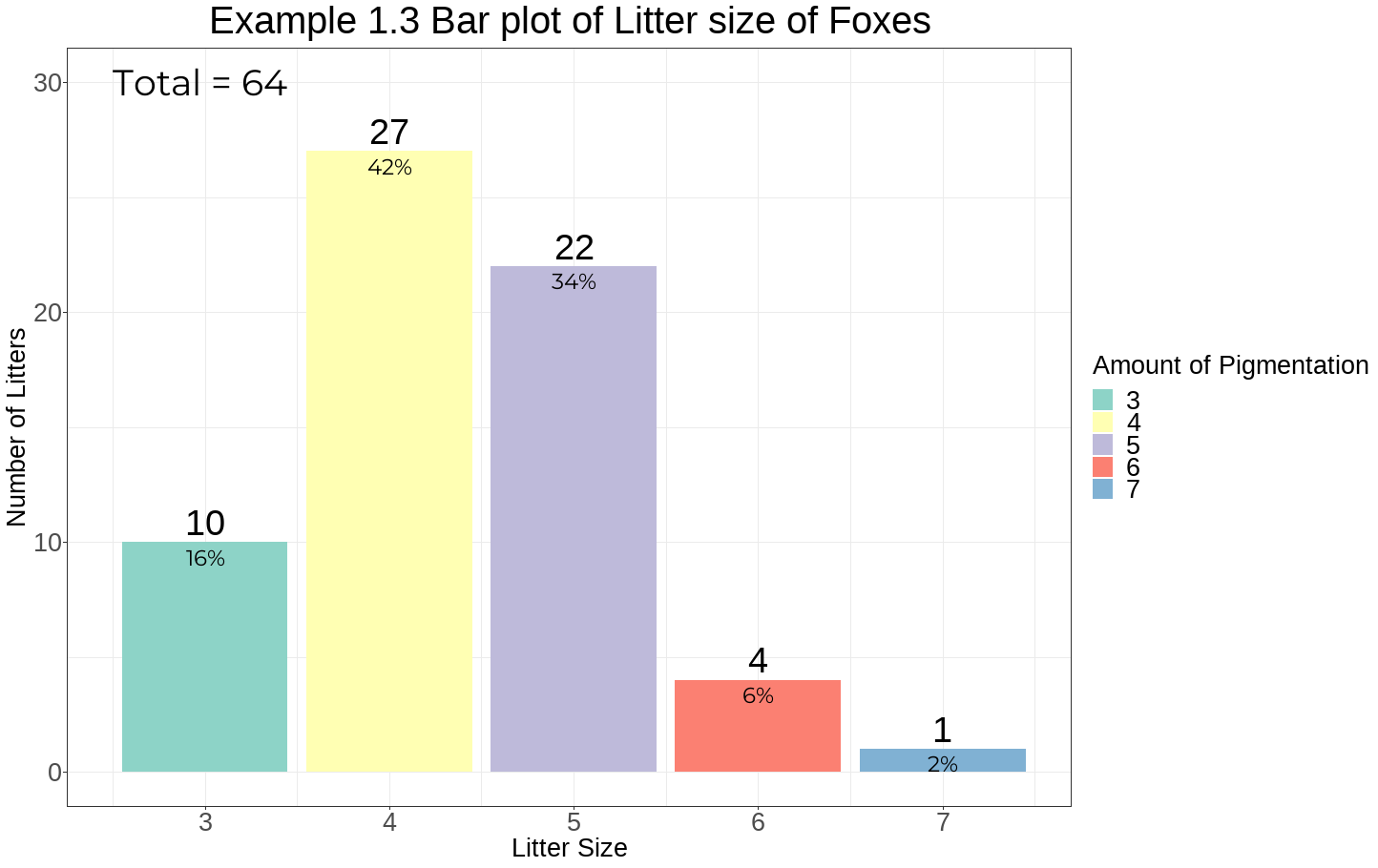
- 총 64마리의 여우를 조사한 결과 한번 출산할 때 네마리를 낳는 경우가 가장 많았으며 그 다음으로 한번에 다섯마리를 낳는 경우가 많게 나왔다.
- 한번에 일곱마리를 낳는 경우가 가장 적게 나타났으며 네마리 이후부터 빈도수가 줄어드는 경향을 보인다.
EXAMPLE 1.4a
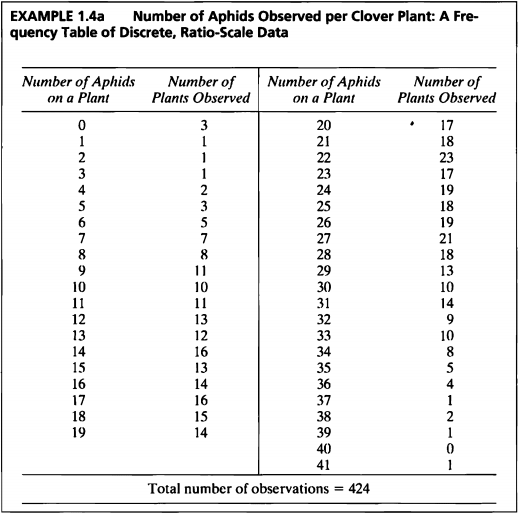
#데이터셋
ex1_4a
## exam1_4a.Aphids exam1_4a.Plants
## 1 0 3
## 2 1 1
## 3 2 1
## 4 3 1
## 5 4 2
## 6 5 3
## 7 6 5
## 8 7 7
## 9 8 8
## 10 9 11
## 11 10 10
## 12 11 11
## 13 12 13
## 14 13 12
## 15 14 16
## 16 15 13
## 17 16 14
## 18 17 16
## 19 18 15
## 20 19 14
## 21 20 17
## 22 21 18
## 23 22 23
## 24 23 17
## 25 24 19
## 26 25 18
## 27 26 19
## 28 27 21
## 29 28 18
## 30 29 13
## 31 30 10
## 32 31 14
## 33 32 9
## 34 33 10
## 35 34 8
## 36 35 5
## 37 36 4
## 38 37 1
## 39 38 2
## 40 39 1
## 41 40 0
## 42 41 1
- 토끼풀 식물에서 관찰된 진딧물 수를 나타낸 데이터이다.
hist1_4a <- ggplot(ex1_4,aes(x=exam1_4a.Aphids,y=exam1_4a.Plants))+
geom_bar(stat="identity",fill="light green")+
theme(plot.title = element_text(hjust = 0.5))+
scale_x_continuous(breaks = seq(0,41,1))+
ggtitle("Example 1.4a Histogram of Number of Aphids observed per Clover plant")+
scale_fill_brewer(palette="Set3")+
ylab("Frequency of Observations")+
xlab("Number of Aphids per plant")+
theme_bw()+
geom_text(aes(label=exam1_4a.Plants),vjust=-0.3,size=7)+
geom_text(aes(label=paste0(round(((exam1_4a.Plants)/sum(exam1_4a.Plants))*100,1),"%")),vjust=2,size=5,family = "raleway")+
ylim(0,25)+
theme(legend.text = element_text(size=20))+
theme(legend.title = element_text(size=20))+
theme(axis.text.x = element_text(size=15))+
theme(axis.text.y = element_text(size=15))+
theme(axis.title = element_text(size=20))+
theme(plot.title = element_text(size=30,hjust = 0.5))+
theme(panel.grid.major.x = element_blank(), panel.grid.minor.x = element_blank(), panel.grid.major.y = element_blank(),
panel.grid.minor.y = element_blank())+
annotate("text", x=2, y=24, label=paste0("Total = ",round(sum(ex1_4$exam1_4a.Plants),2)),family="mont", size=6,hjust=0)+
annotate("text", x=2, y=22, label=paste0("Mean = ",round(mean(ex1_4$exam1_4a.Plants),2)),family="mont", size=6,hjust=0)+
annotate("text", x=2, y=20, label=paste0("STD = ",round(sd(ex1_4$exam1_4a.Plants),2)),family="mont", size=6,hjust=0)+
annotate("rect", xmin = 1.5, xmax = 7, ymin = 19, ymax = 25,alpha = .3,colour="royal blue",fill="skyblue")
hist1_4a
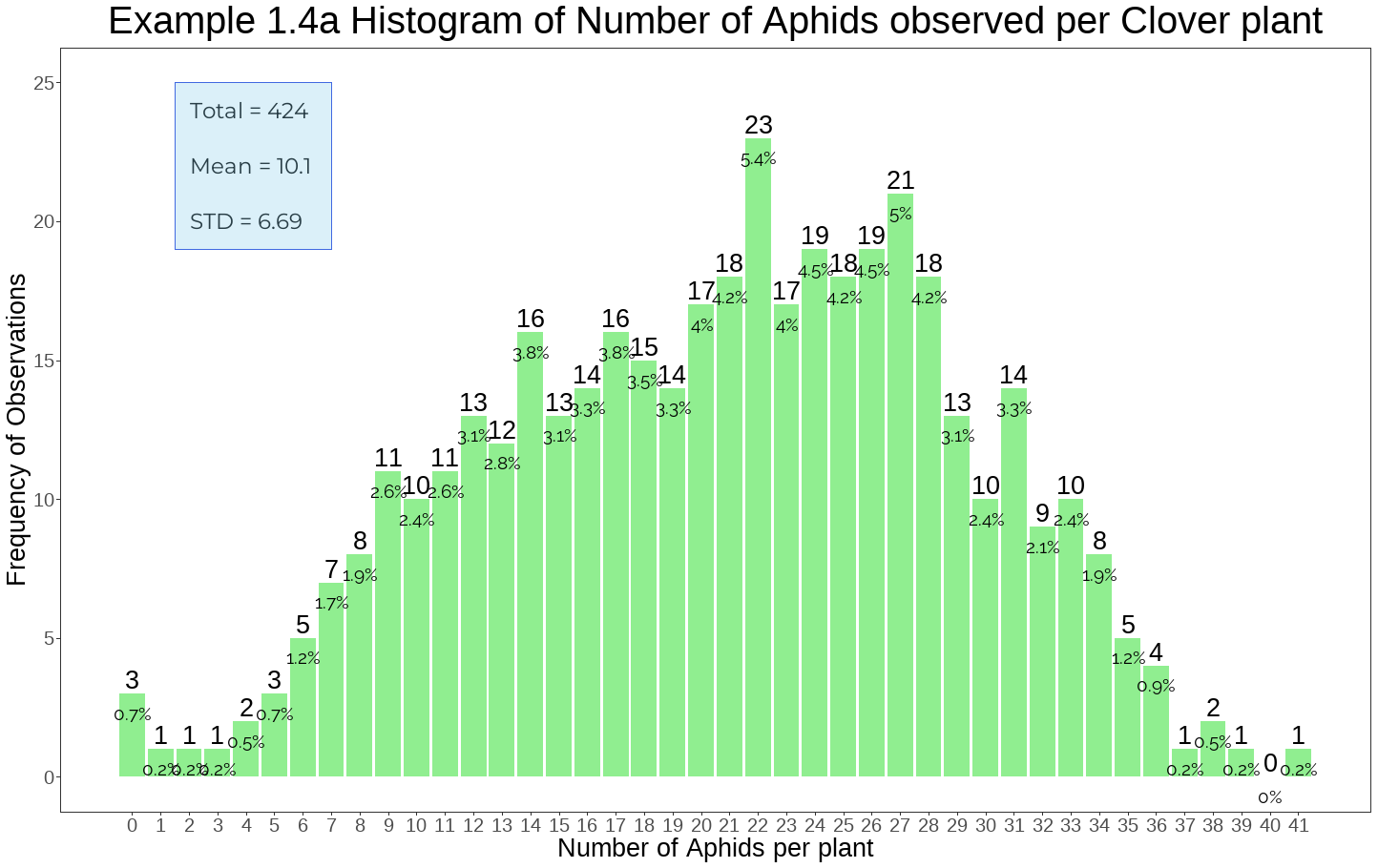
- 그래프의 전체적인 형태를 보면 가운데에 집중되어 있고 끝쪽의 분포가 매우 작게 나타나며 단봉형태를 보인다.
- 토끼풀 식물에서 22마리의 진딧물이 나타난 경우가 가장 많은 것으로 나타나며 20마리에서 30마리 사이의 빈도가 높게 나타나는 것을 볼 수 있다.
- 또한 40마리의 진딧물이 나타난 경우가 없는것으로 나타나며 35마리에서 41마리 사이의 빈도가 낮게 나타나는 것을 볼 수 있다.
- 위와 같이 막대그래프의 경우 너무 길게 나타나는 경우에는 세부적인 정보를 알 수 있으나 그룹화했을 때 그 경향성이 달라질 수 있으므로 그룹화한 그래프 또한 살펴보아야 한다.
EXAMPLE 1.4b
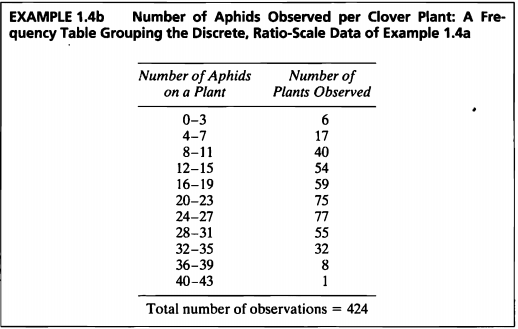
#데이터셋
ex1_4b
## exam1_4b.Aphids exam1_4b.Plants
## 1 0-3 6
## 2 4-7 17
## 3 8-11 40
## 4 12-15 54
## 5 16-19 59
## 6 20-23 75
## 7 24-27 77
## 8 28-31 55
## 9 32-35 32
## 10 36-39 8
## 11 40-43 1
hist1_4b <- ggplot(ex1_4b,aes(x=exam1_4b.Aphids,y=exam1_4b.Plants))+
geom_bar(stat="identity",fill="light green")+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.4b Histogram of Number of Aphids observed per Clover plant")+
scale_fill_brewer(palette="Set2")+
ylab("Frequency of Observations")+
xlab("Number of Aphids per plant")+
theme_bw()+
theme(legend.text = element_text(size=20))+
theme(legend.title = element_text(size=20))+
theme(axis.text.x = element_text(size=15))+
theme(axis.text.y=element_text(size=15))+
theme(axis.title = element_text(size=20))+
theme(plot.title = element_text(size=30,hjust = 0.5))+
theme(panel.grid.major.x = element_blank(), panel.grid.minor.x = element_blank(), panel.grid.major.y = element_blank(),
panel.grid.minor.y = element_blank())+
scale_x_discrete(limits=c("0-3","4-7","8-11","12-15","16-19","20-23","24-27","28-31","32-35","36-39","40-43"))+
geom_text(aes(label=exam1_4b.Plants),vjust=-0.5,size=10)+
geom_text(aes(label=paste0(round(((exam1_4b.Plants)/sum(exam1_4b.Plants))*100,1),"%")),vjust=1.5,size=8,family = "raleway")+
annotate("text", x=1, y=75, label=paste0("Total = ",round(sum(ex1_4b$exam1_4b.Plants),2)),family="mont", size=7,hjust=0)+
annotate("text", x=1, y=71, label=paste0("Mean = ",round(mean(ex1_4b$exam1_4b.Plants),2)),family="mont", size=7,hjust=0)+
annotate("text", x=1, y=67, label=paste0("STD = ",round(sd(ex1_4b$exam1_4b.Plants),2)),family="mont", size=7,hjust=0)+
annotate("rect", xmin = 0.8, xmax = 2.5, ymin = 64, ymax = 78,alpha = .3,colour="royal blue",fill="skyblue")
hist1_4b
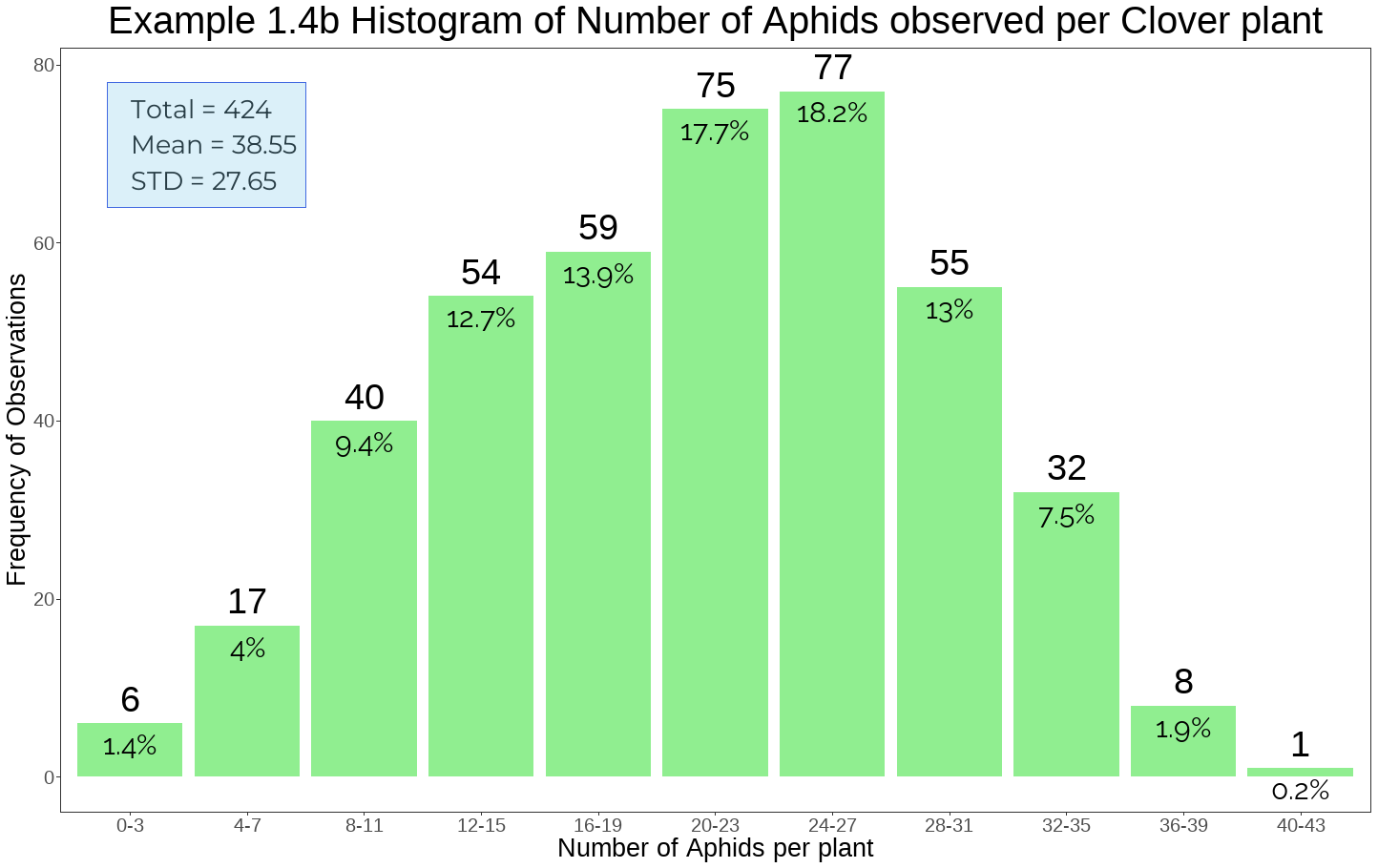
- 토끼풀 식물에서 관찰된 진딧물 수를 동일한 사이즈로 그룹화하여 그 빈도를 나타낸 그래프이다.
- 이 그래프 또한 앞서 살펴본 그래프와 같이 가운데에 집중되어 있고 끝쪽의 분포가 매우 작게 나타나며 단봉형태를 보인다.
- 토끼풀 식물에서 24-27마리의 진딧물이 나타난 경우가 가장 많은 것으로 나타나며 40-43마리의 진딧물이 나타난 경우가 가장 적은 것으로 나타난다.
- 이 경우 앞서 살펴본 그래프와 비슷한 경향성을 띄고 있으나 막대그래프의 경우 너무 길게 나타나면 그래프의 특성을 살리기 어렵고 보기에 다소 불편함이 있으므로 위와 같이 그룹화하여 나타내는 것이 효과적일 수 있다.
EXAMPLE 1.5
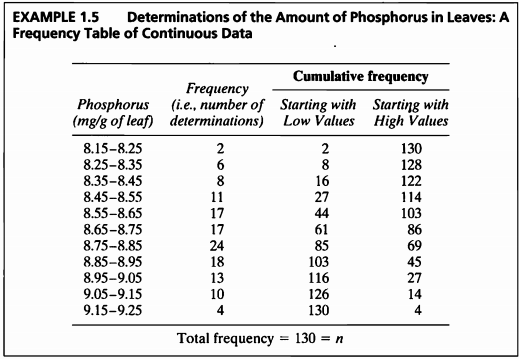
#데이터셋
ex1_5
## exam1_5.Phosphorus exam1_5.Frequency exam1_5.CumFreq1 exam1_5.CumFreq2
## 1 8.15-8.25 2 2 130
## 2 8.25-8.35 6 8 128
## 3 8.35-8.45 8 16 122
## 4 8.45-8.55 11 27 114
## 5 8.55-8.65 17 44 103
## 6 8.65-8.75 17 61 86
## 7 8.75-8.85 24 85 69
## 8 8.85-8.95 18 103 45
## 9 8.95-9.05 13 116 27
## 10 9.05-9.15 10 126 14
## 11 9.15-9.25 4 130 4
- 나뭇잎에서 탐지되는 인의 양에 대한 데이터이다.
phos <- seq(8.2,9.2,0.1)
df <- data.frame(ex1_5, phos)
#plt1 : 그래프를 그리기 위한 데이터와 x,y 축 명시 정보를 가지고 있는 기본 객체, 그리고 각 축에 맞는 이름 설정
plt1 <- ggplot(data=df, aes(x=phos, y=ex1_5$exam1_5.Frequency)) + ylab("Frequency") + xlab("Phosphorus (mg/g of leaf)")
# 객체 plt1 위에 bar graph를 추가, fill 함수는 막대에 색을 채우기 위해 사용
plt1 <- plt1 + geom_bar(stat="identity", fill='#90ee90')
# 업데이트 된 plt1위에 line graph를 추가
plt1 <- plt1 + geom_line(stat="identity")
# 업데이트 된 plt1위에 x 눈금 스케일 정보를 추가
plt1 <- plt1 + scale_x_continuous(breaks=phos)
# plt1 출력
plt1
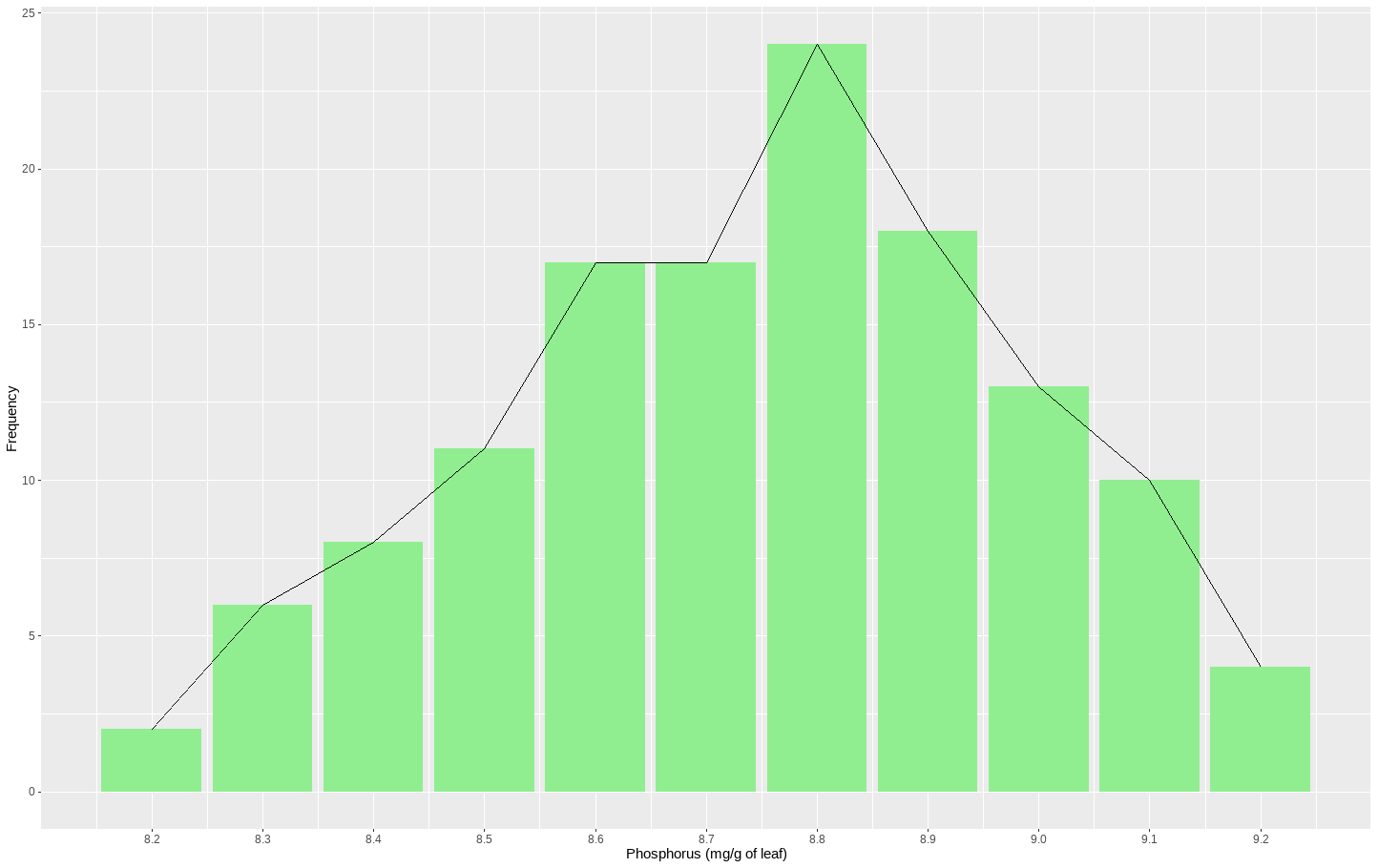
- 히스토그램과 도수다각형을 함께 그려 그래프로 나타낼 경우 히스토그램을 통해서는 자료의 분포를 한눈에 볼 수 있으며 도수다각형 그래프를 통해서는 각 도수에 대한 비교가 용이하며 자료의 경향성을 파악하기 쉽다.
- x축에 나타난 값은 범위로 제공된 인의 양을 대표값(중앙값)으로 나타낸 것이며 8.75-8.85mg에서 가장 높은 빈도를 보였다.
- 가장 높은 빈도를 보인 8.75-8.85mg 기준으로 양쪽 끝으로 갈수록 빈도가 적어지는 단봉형태를 나타내고 있으며 약간 오른쪽으로 치우친 그래프 형태(skewed to the right)를 보이고 있다.
plt2 <- data.frame(phos,ex1_5$exam1_5.Frequency)
plot(plt2,type='b',ylab="Frequency",xlab="Phosphorus(mg/g of leaf)",
ylim=c(0,30), xaxp=c(8.2,9.2,5), pch=19)
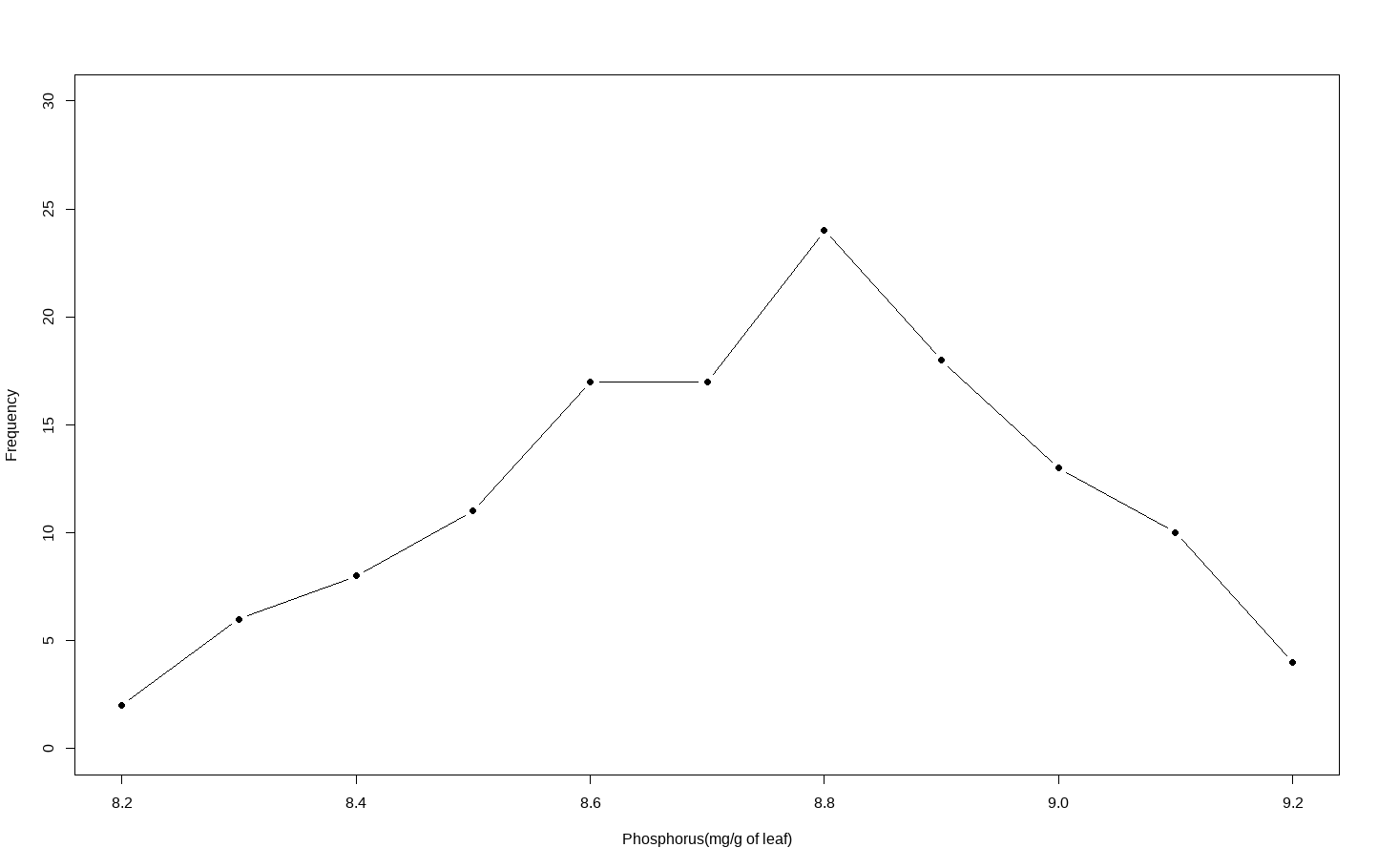
- 다음은 앞서 살펴본 그래프에서 히스토그램을 제외하고 도수다각형만을 나타낸 그래프이다.
- 각각의 값마다 점이 찍혀 있고 그 점을 이은 형태로 도수의 분포를 연속적으로 관찰할 수 있으며 전체적인 그래프의 경향성을 파악하기 쉽다.
hist1_5 <- ggplot(ex1_5,aes(x=exam1_5.Phosphorus,y=exam1_5.Frequency))+
geom_bar(stat = "identity",fill="light green")+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.5 Histogram of leaf phosphrous data")+
scale_fill_brewer(palette="Set2")+
ylab("Frequency")+
xlab("Phosphorus (mg/g of leaf)")+
theme_bw()+
theme(legend.text = element_text(size=20))+
theme(legend.title = element_text(size=20))+
theme(axis.text.x = element_text(size=20))+
theme(axis.text.y=element_text(size=20))+
theme(axis.title = element_text(size=20))+
theme(plot.title = element_text(size=30,hjust = 0.5))+
geom_text(aes(label=exam1_5.Frequency),vjust=-1.5,size=10)+
geom_text(aes(label=paste0(round(((exam1_5.Frequency)/sum(exam1_5.Frequency))*100,1),"%")),hjust=0.3,vjust=3.5,size=8,family = "raleway")+
annotate("text", x=1, y=24, label=paste0("Total = ",round(sum( ex1_5$exam1_5.Frequency),2)),family="mont", size=10,hjust=0,fontface=2)+
annotate("text", x=1, y=22, label=paste0("Mean = ",round(mean( ex1_5$exam1_5.Frequency),2)),family="mont", size=10,hjust=0,fontface=2)+
annotate("text", x=1, y=20, label=paste0("STD = ",round(sd( ex1_5$exam1_5.Frequency),2)),family="mont", size=10,hjust=0,fontface=2)+
annotate("rect", xmin = 1, xmax = 3, ymin = 19, ymax = 25,alpha = .3,colour="tomato",fill="peach puff")+
geom_line(stat="identity",group=1,color="skyblue",size=1.5)+
geom_point(stat="identity",group=1,color="steel blue",size=4)
hist1_5
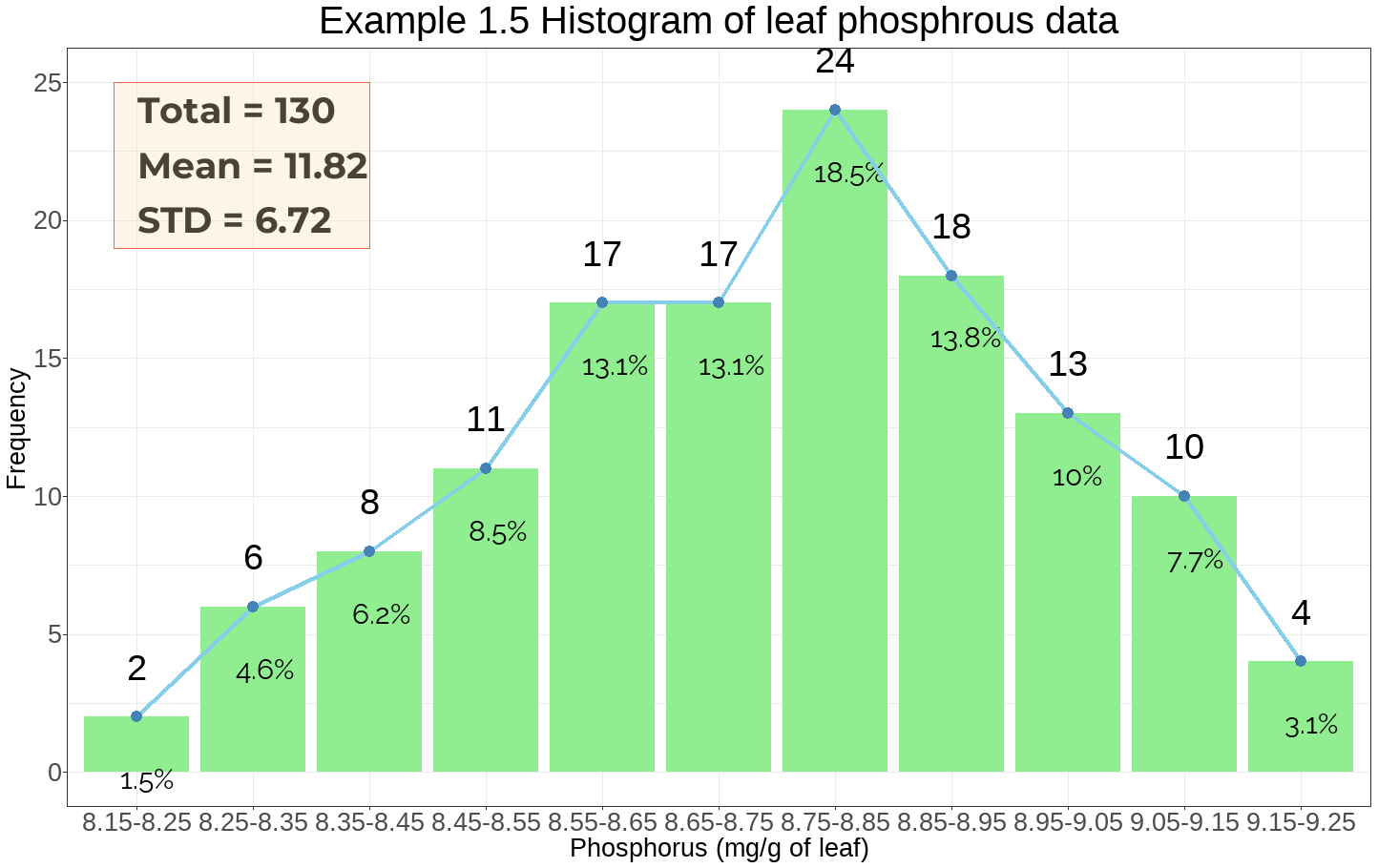
- 위 결과를 보아 인의 양이 8.8인 경우가 24번 관측되었으며 전체 관측수인 130중 18.5%를 차지한다.
- 관측수에 대한 평균은 11.82이며 표준편차는 6.72이다.
cum1 <- ggplot(ex1_5,aes(x=plt2$phos,y=ex1_5$exam1_5.CumFreq1))+
scale_x_continuous(breaks = plt2$pho)+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.5 Cumulative Frequency Polygon of leaf phosphrous data")+
scale_fill_brewer(palette="Set2")+
ylab("Cumulative Frequency")+
xlab("Phosphorus (mg/g of leaf)")+
theme_bw()+
theme(legend.text = element_text(size=15))+
theme(legend.title = element_text(size=15))+
theme(axis.text.x = element_text(size=10))+
theme(axis.text.y=element_text(size=10))+
theme(axis.title = element_text(size=15))+
theme(plot.title = element_text(size=20,hjust = 0.5))+
geom_text(aes(label=exam1_5.CumFreq1),hjust=1,vjust=-1,size=4)+
geom_text(aes(label=paste0(round((exam1_5.CumFreq1/max(exam1_5.CumFreq1))*100,1),"%")),hjust=0.2,vjust=2,size=4,family = "raleway")+
geom_line(stat="identity",group=1,color="light green",size=3)+
geom_point(stat="identity",group=1,color="coral",size=5)+
scale_y_continuous(breaks = seq(0, 140, 20), sec.axis = sec_axis( ~./max(ex1_5$exam1_5.CumFreq1),name = "Relative Cumulative Frequency"))
cum1
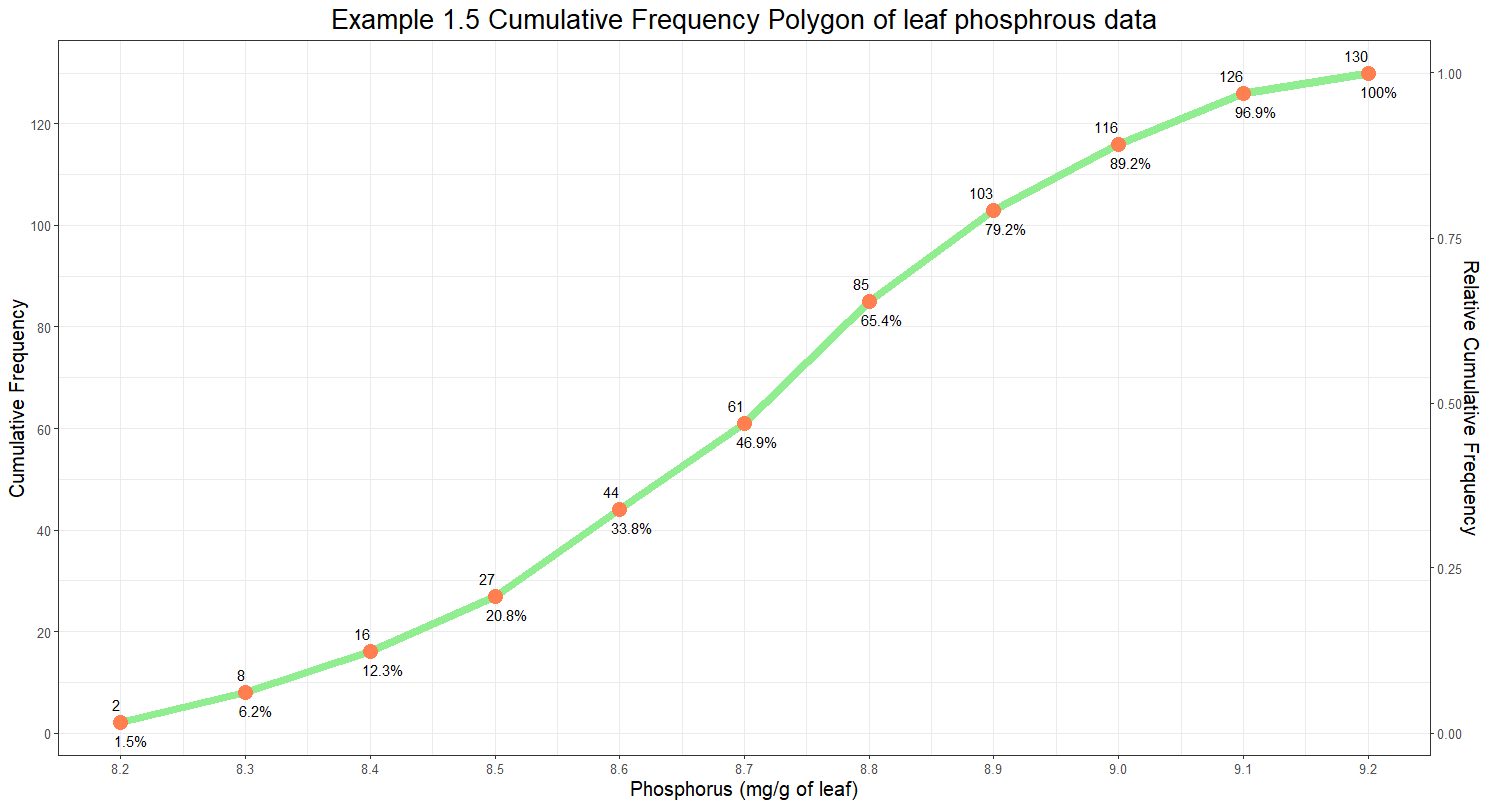
- 나뭇잎에서 탐지되는 인의 양을 누적도수다각형으로 나타낸 그래프이다.
- 누적그래프이므로 상대도수의 마지막 값이 1인것을 볼 수 있으며 그래프의 전체적인 형태는 S자 형태를 보인다.
- 앞서 살펴본 히스토그램과 도수다각형을 통해 알 수 있었던 것처럼 양끝으로 갈수록 그래프의 증가 폭이 작아지는 것을 통해 양 극단의 도수가 작고 중간 값의 도수가 크다는 것을 알 수 있다.
cum2 <- ggplot(ex1_5,aes(x=plt2$phos,y=exam1_5.CumFreq2))+
scale_x_continuous(breaks = plt2$pho)+
theme(plot.title = element_text(hjust = 0.5))+
ggtitle("Example 1.5 Cumulative Frequency Polygon of leaf phosphrous data")+
scale_fill_brewer(palette="Set2")+
ylab("Cumulative Frequency")+
xlab("Phosphorus (mg/g of leaf)")+
theme_bw()+
theme(legend.text = element_text(size=15))+
theme(legend.title = element_text(size=15))+
theme(axis.text.x = element_text(size=10))+
theme(axis.text.y=element_text(size=10))+
theme(axis.title = element_text(size=15))+
theme(plot.title = element_text(size=20,hjust = 0.5))+
geom_text(aes(label=exam1_5.CumFreq2),hjust=0,vjust=-1,size=4)+
geom_text(aes(label=paste0(round(((exam1_5.CumFreq2)/max(exam1_5.CumFreq1))*100,1),"%")),hjust=1,vjust=2,size=4,family = "raleway")+
geom_line(stat="identity",group=1,color="light green",size=3)+
geom_point(stat="identity",group=1,color="coral",size=5)+
scale_y_continuous(breaks = seq(0, 140, 20), sec.axis = sec_axis( ~./max(ex1_5$exam1_5.CumFreq1),name = "Relative Cumulative Frequency"))
cum2
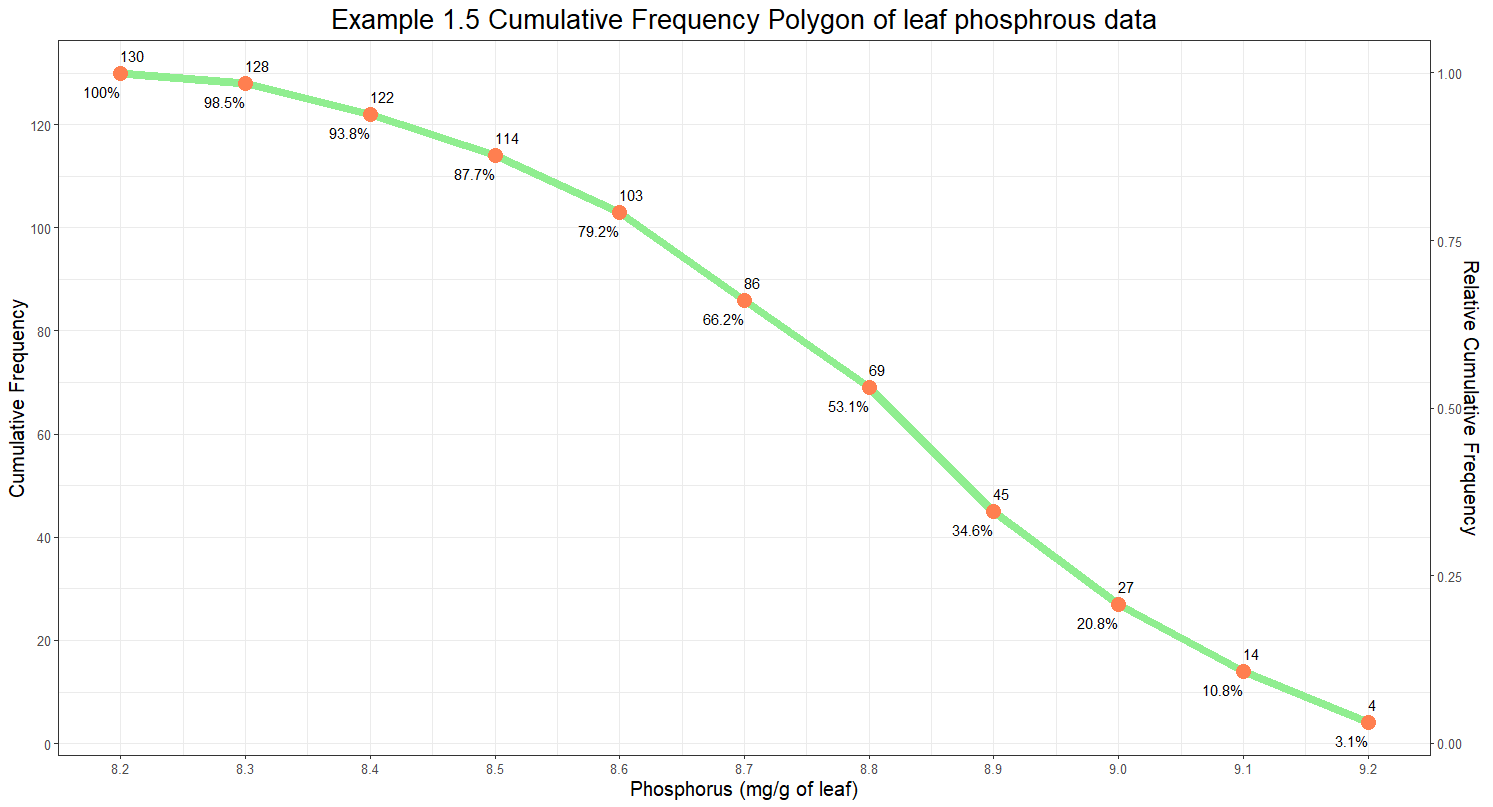
- 앞서 살펴본 그래프의 누적도수를 역순으로 하여 나타낸 그래프이다.
SAS 프로그램 결과
SAS 접기/펼치기 버튼
1장
1장 연습문제 불러오기
LIBNAME ex 'C:\Biostat';
RUN;
/*1장 연습문제 불러오기*/
%macro chap01_1(name=,no=);
%do i=1 %to &no.;
PROC IMPORT DBMS=excel
DATAFILE="C:\Biostat\data_chap01"
OUT=ex.&name.&i. REPLACE;
RANGE="exam1_&i.$";
RUN;
%end;
%mend;
%macro chap01_2(name=,no=);
PROC IMPORT DBMS=excel
DATAFILE="C:\Biostat\data_chap01"
OUT=ex.&name.&no. REPLACE;
RANGE="exam1_&no.$";
RUN;
%mend;
%chap01_1(name=ex1_,no=5);
%chap01_2(name=ex1_,no=4a);
%chap01_2(name=ex1_,no=4b);
EXAMPLE 1.1
/*둥지 개수를 bar graph로 출력하기*/
title 'The Location of Sparrow Ners';
/*bar plot1*/
PROC SGPLOT DATA=ex.ex1_1;
styleattrs datacolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3") datacontrastcolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3");
vbarparm category=nestsite response=number / group=nestsite;
yaxis label="Number of Nests" ;
xaxis label="Nest Site";
RUN;
/*bar plot2 : 세로축이 45부터 시작되는 그래프*/
PROC SGPLOT DATA=ex.ex1_1;
styleattrs datacolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3") datacontrastcolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3");
vbarparm category=nestsite response=number / group=nestsite;
yaxis grid values=(45 to 60 by 5)
label="Number of Nests" labelattrs=(size=7pt);
xaxis label="Net Site";
RUN;
EXAMPLE 1.2
/*색소 침착 빈도를 bar graph로 출력하기*/
title 'Amont of Black Pigmentation';
PROC FREQ DATA=ex.ex1_2;
weight number;
tables class / out=ex.ex1_2out;
RUN;
/*bar plot*/
PROC SGPLOT DATA=ex.ex1_2out;
styleattrs datacolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3") datacontrastcolors=('#fb8072' "#8dd3c7" "#bebada" "#ffffb3" "#80b1d3");
vbarparm category=class response=count / group=class;
xaxistable count / colorgroup=class location=inside nolabel;
xaxis label="Pigmentation class";
yaxis label="Number of fish" grid values= (0 to 70 by 10);
RUN;
FREQ 프로시저
| Class | ||||
|---|---|---|---|---|
| Class | 빈도 | 백분율 | 누적 빈도 | 누적 백분율 |
| 0 | 13 | 8.44 | 13 | 8.44 |
| 1 | 68 | 44.16 | 81 | 52.60 |
| 2 | 44 | 28.57 | 125 | 81.17 |
| 3 | 21 | 13.64 | 146 | 94.81 |
| 4 | 8 | 5.19 | 154 | 100.00 |
EXAMPLE 1.3
PROC SGPLOT DATA=ex.ex1_3;
styleattrs datacolors=("#8dd3c7" "#ffffb3" "#bebada" '#fb8072'"#80b1d3") datacontrastcolors=("#8dd3c7" "#ffffb3" "#bebada" '#fb8072'"#80b1d3");
vbarparm category=size response=frequency / group=size;
xaxistable frequency /location=inside nolabel;
yaxis label="Number of Litters" values=(0 to 30 by 5) valueshint;
xaxis label="Litter Size";
RUN;
EXAMPLE 1.4a
/*클로버당 관찰된 진딧물의 수의 빈도를 그래프로 나타내었다.*/
PROC SGPLOT DATA=ex.ex1_4a;
vbarparm category=aphids response=plants / group=aphids;
xaxistable plants / colorgroup=aphids location=inside nolabel;
xaxis label="Observed Number of Aphids per Plant";
yaxis label="Frequency of Observations";
RUN;
EXAMPLE 1.4b
PROC FREQ DATA=ex.ex1_4b;
TABLES aphids;
WEIGHT plants;
RUN;
PROC SGPLOT DATA=ex.ex1_4b;
vbarparm category=aphids response=plants / group=aphids;
xaxistable plants / colorgroup=aphids location=inside nolabel;
xaxis label="Observed Number of Aphids per Plant";
yaxis label="Frequency of Observations" grid values= (0 to 80 by 10);
RUN;
FREQ 프로시저
| Aphids | ||||
|---|---|---|---|---|
| Aphids | 빈도 | 백분율 | 누적 빈도 | 누적 백분율 |
| 0-3 | 6 | 1.42 | 6 | 1.42 |
| 12-15 | 54 | 12.74 | 60 | 14.15 |
| 16-19 | 59 | 13.92 | 119 | 28.07 |
| 20-23 | 75 | 17.69 | 194 | 45.75 |
| 24-27 | 77 | 18.16 | 271 | 63.92 |
| 28-31 | 55 | 12.97 | 326 | 76.89 |
| 32-35 | 32 | 7.55 | 358 | 84.43 |
| 36-39 | 8 | 1.89 | 366 | 86.32 |
| 4-7 | 17 | 4.01 | 383 | 90.33 |
| 40-43 | 1 | 0.24 | 384 | 90.57 |
| 8-11 | 40 | 9.43 | 424 | 100.00 |
EXAMPLE 1.5
PROC SGPLOT DATA=ex.ex1_5_phos;
vbar real_phosphorus / response = Frequency barwidth=1;
xaxis label= "Phosphorus (mg/g of leaf)" ;
yaxis label="Frequency" values=(0 to 30 by 5) ;
TITLE 'Determinations of the Amount of Phosphorus in Leaves';
RUN;
DATA ex.ex1_5_phos;
set ex.ex1_5;
lower = substr(Phosphorus, 1,4)*1;
upper= substr(Phosphorus, 6, 6)*1;
real_phosphorus = mean(lower,upper);
RF = Frequency / 130;
RCF = CumFreq1 / 130;
RCF2 = CumFreq2 / 130;
RUN;
PROC SGPLOT DATA=ex.ex1_5_phos;
keylegend / title=" ";
series x=real_phosphorus y=RF / y2axis transparency=1;
series x=real_phosphorus y=Frequency / markers ;
xaxis label="Phosphorus (mg/g of leaf)" values=(8.2 to 9.2 by 0.1);
yaxis label="Frequency" values=(0 to 30 by 5);
y2axis label="Relative Frequency" values= (0 to 0.2 by 0.05);
RUN;
PROC SGPLOT DATA=ex.ex1_5_phos;
keylegend / title=" ";
series x=real_phosphorus y=RCF / y2axis transparency=1;
series x=real_phosphorus y=CumFreq1 / markers ;
xaxis label="Phosphorus (mg/g of leaf)" values=(8.2 to 9.2 by 0.1);
yaxis label="Cumulative Frequency" values=(0 to 130 by 10) ;
y2axis label="Relative Cumulative Frequency" values=(0 to 1 by 0.2);
RUN;
PROC SGPLOT DATA=ex.ex1_5_phos;
keylegend / title=" ";
series x=real_phosphorus y=RCF2 / y2axis transparency=1;
series x=real_phosphorus y=CumFreq2 / markers ;
xaxis label="Phosphorus (mg/g of leaf)" values=(8.2 to 9.2 by 0.1);
yaxis label="Cumulative Frequency" values=(0 to 130 by 10) ;
y2axis label="Relative Cumulative Frequency" values=(0 to 1 by 0.2);
RUN;
교재: Biostatistical Analysis (5th Edition) by Jerrold H. Zar
**이 글은 22학년도 1학기 의학통계방법론 과제 자료들을 정리한 글 입니다.**
